丙酮酸脱氢酶复合物-酶2,是李斯特菌的新靶标。结合噬菌体展示技术确定的检测
丙酮酸脱氢酶复合物-酶2,是李斯特菌的新靶标。结合噬菌体展示技术确定的检测
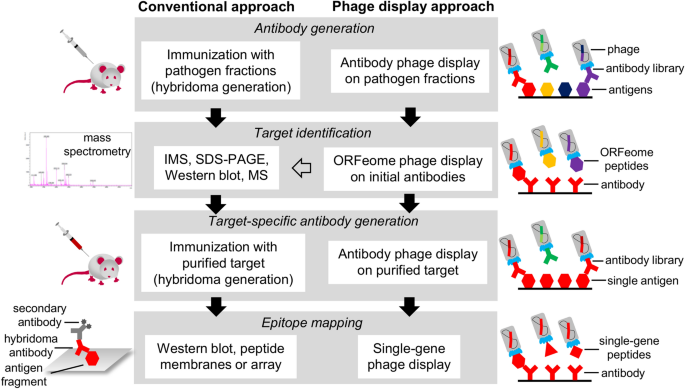
属李斯特菌包括无处不在的细菌,通常存在于食品和食品生产设施。在这项研究中,采用了三种不同的噬菌体展示技术来发现靶标,并生成和表征针对李斯特菌的新型抗体:用于生物标志物发现和抗体生成的抗体展示;或用于目标识别的信息显示;以及用于表位表征的单基因展示。通过这种方法,丙酮酸脱氢酶复合物酶2(PDC-E2)被定义为李斯特菌的新检测目标由免疫磁分离质谱法(IMS-MS)确认。免疫印迹和荧光显微镜检查显示该蛋白可在活细胞的细菌细胞表面上接近。重组PDC-E2在大肠杆菌中产生,并用于产生16种其他抗体。在间接ELISA中针对17个李斯特菌和16个非李斯特菌物种测试了所得的一组20个单克隆scFv-Fc 。其中两个提供了100%的敏感性(CI 82.35–100.0%)和特异性(CI 78.20–100.0%),确认PDC-E2是检测李斯特菌的合适靶点。分析了这些抗体中18种的结合区域,发现约90%(16/18)与靶标的脂酰结构域(LD)结合。新型靶标PDC-E2和针对它的高度特异性抗体为改善李斯特菌的检测提供了新的机会。
介绍
属李斯特菌包括革兰氏阳性,兼性厌氧菌,非孢子,棒状的细菌。有6个李斯特菌,直到2009年描述的物种,从该最后的种类在1984年描述1。后来,14新种进行了说明2,3,4,5,6,7,8,导致总共20:李斯特菌,L. ivanovii,无害利斯特氏菌,L. seeligeri,L. welshimeri,L. grayi,L. marthii,L. rocourtiae,L. fleischmannii,L. weihenstephanensis,L. booriae,L. newyorkensis,L. floridensis,L.菜类,L. cornellensis,L.河岸,L. grandensis,L. goaenensis,L. costaricensis,和大号。thailandensis。近年来,如此大量的新物种反映出人们对李斯特菌属作为重要研究对象的认识不断提高。与人类最相关的病原体仍然是单核细胞增生李斯特菌,而伊万诺维氏杆菌对兽医更重要9。然而,单增李斯特菌含有从基因菌株L. ivanovii已被描述为是高毒性,增强这两个物种的重要性10。目前,已经描述了13种不同的单核细胞增生李斯特菌血清型,其中3种(4b,1 / 2a和1 / 2b)被认为是最强毒力的,占人类感染的95-98%11。
在李斯特菌属是无处不在的,它可以在环境中的许多地方找到,以及在动物和人类的微生物。此外,它对宽范围的pH和温度,高浓度的盐和低含量的氧气具有较高的抵抗力,并且具有形成生物膜的能力。这些特性有助于食品生产设施的持续存在12,13,14。结合其复杂的肠道入侵机制,使单核细胞增生李斯特菌重大的食源性病原体。由这些细菌引起的疾病被称为李斯特菌病,主要影响免疫功能低下的人,例如移植,癌症和HIV患者,以及婴儿,老人,孕妇及其胎儿。尽管李斯特菌病的发病率很低(在大多数国家中不到100,000人),但它仍然是一个严重的威胁,因为全世界的死亡率约为25%15。因此,在美国等国家/地区,要求即食(RTE)食品中的李斯特菌必须 “零容忍”的法律14。相比之下,在欧洲许多地方,法律并不严格,允许RTE食品中每克食品最高100 CFU / g 16。在其他国家,关于某些高风险食品的立法更少或没有,使得李斯特菌污染成为更大的问题。因此,在生产的食品或食品生产设施中检测李斯特菌至关重要。在食品中对其进行检测的标准方法涉及冗长的微生物程序,以进行富集,分离和生化表征17。尽管非常具体和精确,但是从样品收集到结果的整个过程大约需要7天。作为替代方案,已开发出诸如基于PCR的技术,免疫学方法,蛋白质组学方法和不同形式的生物传感器等程序,以将检测时间减少到大约1天或更短18。
在这些技术中,基于抗体的检测方法(例如侧流检测)被认为是最有前途的,因为它们提供了一种简单,快速且低成本的检测方法。尽管对食品工业非常有吸引力,但是横向流测定以及其他新方法仍然取决于检测之前的有效富集步骤,以增加样品中可检测细胞的数量。在富集步骤中已实现了显着改善,大大减少了获得可检测量细菌细胞的时间19。在另一项研究中,通过改进比色成分20可以成功地提高对侧向流的检测极限。。但是,用于侧向流动测定的合适的和特异性的单克隆抗体(mAb)的开发并没有以相同的方式进行,从而有待改进。
大多数用于研究检测目标的利斯特氏菌都涉及到检测单增李斯特菌,如internalins A(INLA)和B(InlB)21,22。这些蛋白质参与肠道入侵步骤的发病机理,并且可以通过附着在细胞壁上在细胞表面上接近。除了这两种蛋白质,其他人也用于检测病原性物种,例如肌动蛋白聚合蛋白(ACTA)的23,或ñ -乙酰基胞壁质酶24,25。不仅检测病原体很重要,而且对非致病性李斯特菌的识别也很重要的,因为它已经表明,非病原性物种能够在富集步骤长满致病,因此增加了生产在检测假阴性结果的几率26,27,28。在这方面,已经描述了几种靶标,例如p60 29,鞭毛蛋白30或1,6-果糖双磷酸醛缩酶(FBA)31,用于检测病原性和非病原性李斯特菌。
噬菌体展示是一种用途广泛的技术,已广泛用于研究抗体和抗原。作为一种显示方法,这种方法将基因型和表型联系起来,从而更易于访问从相互作用过程32中选择的分子的遗传信息。通过一种称为淘选的方法(称为“淘金”),可以选择具有所需相互作用特性的分子。通过噬菌体展示产生的重组mAbs目前用于基础研究,诊断和治疗应用的有价值的资源33,34,35,36,37。在另一方面,在抗原噬菌体展示技术的改进已经实现,由于向Hyperphage的可用性38如ORFeome噬菌体展示,39,40,41。该技术可用于发现生物标志物,然后可用于开发疫苗和诊断方法。最后,单基因噬菌体展示可通过鉴定“最小识别序列”(MSR)来表征抗体结合的抗原区域,“最小识别序列”表示存在于每个输出序列中的抗原的最短氨基酸片段。对准之后的步骤42,43,44。
本研究表明,结合使用的噬菌体展示技术可以获取用于检测的生物标记,针对它的抗体以及相关的表位信息。在这方面,产生了针对新鉴定的靶标二氢脂酰胺乙酰转移酶(丙酮酸脱氢酶复合物-酶2,PDC-E2)的新型重组单克隆抗体,然后用于表征。此外,这些抗体可以特异性检测包括在研究中的所有17 种李斯特菌。
结果
李斯特菌蛋白级分的抗体淘选为活细胞检测提供了结合剂
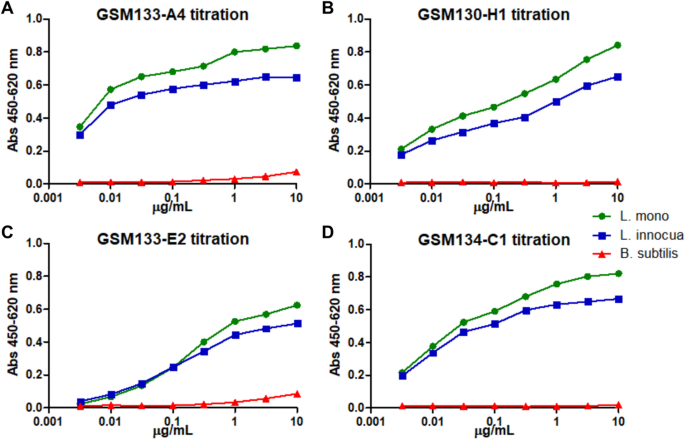
为了避免使用全细胞,使用了利斯特氏菌的细胞壁,膜和细胞质的亚细胞蛋白级分,因为它们在淘选过程中倾向于从聚苯乙烯表面分离。另外,它增加了获得抗体的机会,这些抗体主要来自细胞壁和细胞膜部分,可以抵抗暴露的蛋白质。这样,通过噬菌体展示产生了针对李斯特菌蛋白级分的scFv格式的抗体(图S1),并转化为scFv-Fc格式(具有小鼠IgG2a Fc)。所得的四种抗体对活细菌细胞表现出浓度依赖性结合,可检测致病性(单核细胞增生李斯特菌)和非致病性(无毒李斯特菌)。)利斯特氏菌属种,但与枯草芽孢杆菌无明显反应用作阴性对照(图 1)。尽管无法精确确定EC 50,但GSM133-A4在稀释度排名中显示出最佳结合,因此选择该抗体用于靶标的进一步初始表征。
通过质谱或ORFeome噬菌体展示进行目标识别可提供相似的结果
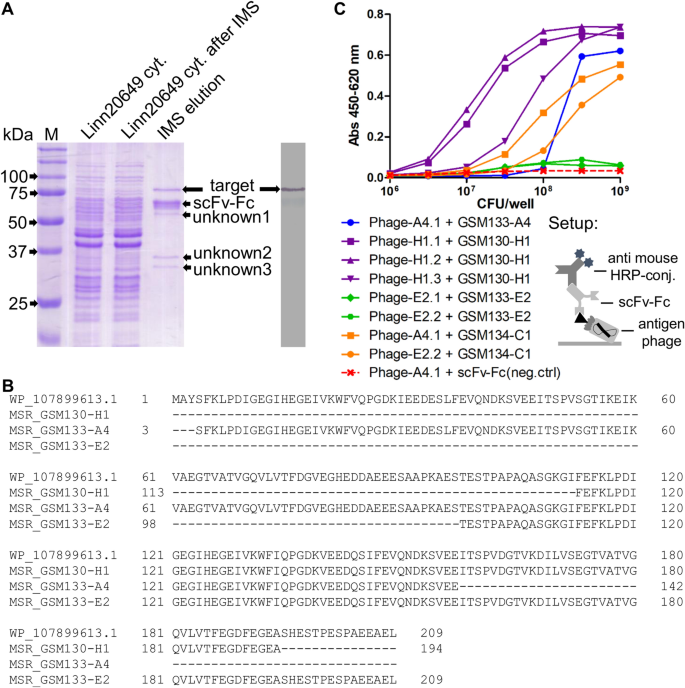
产生针对利斯特氏菌的蛋白质级分的抗体后,可通过IMS-MS(免疫磁分离质谱法)或ORFeome噬菌体展示进一步确定其靶标。在MS分析之前,使用GSM133-A4制备具有蛋白A珠的IMS样品,以从细胞质蛋白混合物中分离靶标。此过程导致了目标的适当富集,通过SDS-PAGE和免疫印迹(图所证实 2 A)。除靶标外,还在SDS-PAGE中检测到其他三个蛋白带(称为“未知” 1-3),但在免疫印迹中未检测到。从凝胶上切下所有蛋白质,并通过MS进行分析(表S1),将目标物识别为二氢脂酰胺乙酰转移酶(GenBank:WP_010990728.1),也称为丙酮酸脱氢酶复合物酶2(PDC-E2)。“未知”蛋白2和3分别检测为PDC-E1α(GenBank:WP_072572643.1)和PDC-E1β(GenBank:WP_038409535.1)。该信息表明“未知1”蛋白可能是指PDC-E3,但由于其与scFv-Fc极为接近而无法正确分析。
通过MS和ORFeome噬菌体展示鉴定目标。(A)在IMS之后用GSM133-A4 scFv-Fc的SDS-PAGE和免疫印迹;M是蛋白质梯形Precision Plus蛋白质未染色(Bio-Rad)。(B)在ORFeome噬菌体展示后,参考PDC-E2序列(WP_107899613.1)的前209个氨基酸与scFv-Fc GSM130-H1,GSM133-A4和GSM133-E2的MSR比对。(C)4种初始scFv-Fc [GSM133-A4(蓝色),GSM130-H1(紫色),GSM133-E2(绿色)和GSM134-C1(黄色)]的滴定曲线对不同量的单克隆噬菌体的分离ORFeome噬菌体显示可用于目标识别。分析的设置显示在图形下方。完整的SDS-PAGE和免疫印迹图像可以分别作为补充图找到。S7和S8。
与MS分析同时,使用构建有L基因组的抗原库进行ORFeome噬菌体展示,以鉴定四种初始抗体(GSM130-H1,GSM133-A4,GSM133-E2和GSM134-C1)的靶标。单核细胞增生性细菌ATCC7644。通过这种方法,鉴定出的靶标也是PDC-E2(GenBank:WP_107899613.1)(图 2 B,表S1)。在使用的四种抗体中,只有GSM134-C1不允许识别目标。此外,用GSM133-A4淘选导致两个结果:一个涉及PDC-E2,另一个涉及由于低反应性而被丢弃的无关蛋白(数据未显示)。
为了证实特异性结合,产生了PDC-E2肽噬菌体的子集,并在ELISA中对各个抗体进行了测试。与阴性对照相比,所有抗体-肽噬菌体组合均以浓度依赖性方式显示出更高的反应(图 2 C)。此外,由于3种抗体的分离片段在蛋白质序列上彼此密切相关,因此使用GSM134-C1测试了其中的一些片段,该片段在淘选时未选择任何片段。结果表明,该scFv-Fc可以识别通过淘选而与其他抗体分离的肽,表明它可以结合相似的表位区域。此外,重要的是要提到GSM133-E2的反应远低于其他抗体,表明该scFv-Fc的行为不同。
PDC-E2存在于李斯特菌细胞壁蛋白组分中,并且在其表面上可及
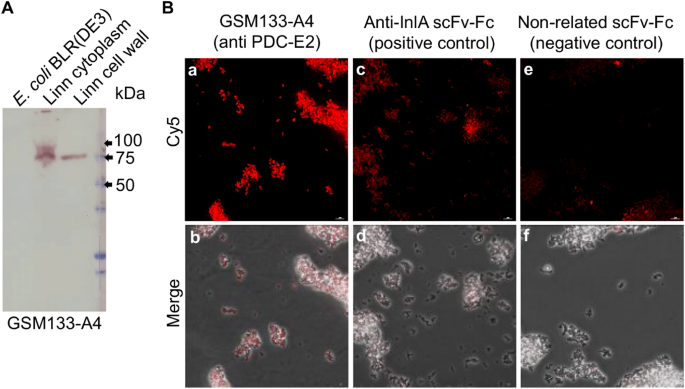
为了分析鉴定的靶标PDC-E2的发生情况,该靶标PDC-E2是预期位于细胞质中的蛋白,在不同的李斯特菌细胞区室中,将抗体GSM133-A4用于李斯特菌属细胞质和细胞壁蛋白组分的免疫印迹试验(图 3)一个)。结果表明,靶抗原存在于细胞质以及李斯特菌细胞的细胞壁上。这与全细胞ELISA中GSM133-A4的检测一致。
李斯特菌蛋白级分的免疫印迹和免疫荧光显微镜检查中的GSM133-A4测试。(甲)从细胞质和细胞壁蛋白质级分的GSM133-A4的免疫印迹无害利斯特氏菌 DSM20649(属),以及大肠杆菌 BLR(DE3)用作阴性对照提取物。印迹显示,GSM133-A4抗体与利斯特氏菌的细胞质和细胞壁组分中所含的靶标结合。所用的蛋白质阶梯是Precision Plus Protein All Blue(Bio-Rad)。(B)活的单核细胞增生李斯特菌的荧光显微镜检查4b DSM15675。GSM133-A4的信号(c和f)显示高于不相关抗体的阴性对照(a和d)和抗InlA阳性对照(b和e)。完整的免疫印迹图像可参见补充图S9。
为了进一步确认PDC-E2在细菌表面的可及性,采用GSM133-A4分析单核细胞增生李斯特菌 ser。图4b DSM 15675通过免疫荧光显微术(图 3 B,A,B)。作为阴性对照,使用非相关的scFv-Fc来验证第二抗体的非特异性结合。阴性对照显示出难以察觉的荧光信号(图 3 B,c,d),而使用scFv-Fc抗InlA抗体的阳性对照则识别出其特征明确的靶标,该靶标与单核细胞增生李斯特菌表面上的细胞壁相连(图 3 B,E,F)。相比之下,GSM133-A4染色显示出更高的信号,表明利斯特氏菌可接近靶标 细胞表面。
重组PDC-E2上的抗体淘选增加了李斯特菌的结合物数量。检测
为了增加识别PDC-E2的抗体组,对纯化的重组抗原进行了另一次抗体淘选。克隆了PDC-E2基因,并在大肠杆菌中表达为重组蛋白。纯化后,将其用于从HAL9和HAL10文库中选择抗体,将它们混合在同一淘选孔中。用这种方法,鉴定了16种新颖的结合物,并产生了具有鼠Fc部分的scFv-Fc。初步测试表明,它们都能够在免疫印迹中识别重组PDC-E2,并特异性结合李斯特菌。在ELISA中(图S2和S3)。
PDC-E2允许检测李斯特菌。通过间接ELISA
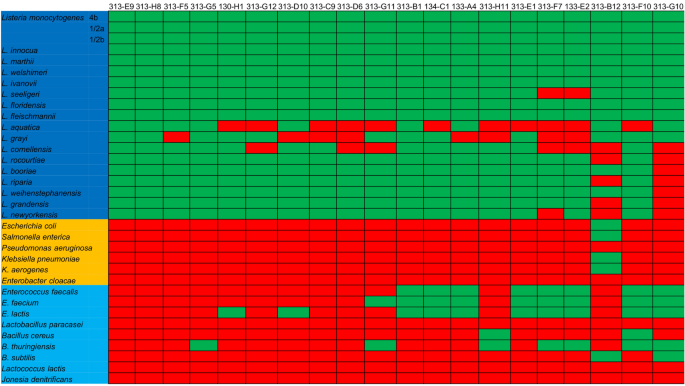
总共测试了20种单克隆scFv-Fc抗体的李斯特菌。间接ELISA检测。进行ROC(接收器工作特性)分析后,使用显示最佳AUC(曲线下面积)的浓度来估算灵敏度和特异性的最佳平衡。允许使用两个scFv-Fc(GSM313-E9和GSM313-H8)来区分李斯特菌。来自所有其他测试物种的灵敏度和特异性为100%,AUC = 1.0(表1)。性能次佳的三种抗体是GSM313-F5,GSM313-G12和GSM130-H1,其中GSM130-H1是唯一从李斯特菌蛋白级分最初淘选而来的抗体。
当针对测试菌株分析反应时,观察到一些明显的识别模式。首先,物种L.菜类,L. grayi,和L. cornellensis难以被检测到,因为出20种抗体,11,图8和7,分别不能够识别它们(图 4)。另一个观察结果是,与非李斯特氏菌属的大多数反应都针对肠球菌(11/20)和芽孢杆菌(8/20)属,有时每个属中有多个属。有趣的是,来自肠球菌和芽孢杆菌的 PDC-E2序列这些是与单核细胞增生李斯特氏菌的亲缘关系更近的物种,具有约50-60%的同一性,而除副干酪乳杆菌(55.7%)以外的其他物种未超过45%(表S2)。
大多数产生的抗体结合到PDC-E2的脂酰结构域
为了更详细地分析抗PDC-E2抗体的结合特性,使用展示PDC-E2随机片段的文库通过单基因噬菌体展示确定了其MSR(最小识别序列)。可以为20种抗体中的18种定义MSR。有趣的是,除一个映射的抗体外,所有抗体都显示出结合同一区域,该区域是第一或第二脂酰结构域(LD)(表2)。有趣的是,在18种抗体中,有4种(GSM133-A4,GSM313-D10,GSM313-F7和GSM313-G10)能够识别两个LD域,尽管始终首选一个。GSM133-E2,最初显示出非常低的反应(图 2C)显示结合催化结构域(最初未被ORFeome展示定义的MSR)和一个无义肽(翻译的肽不对应于PDC-E2)。因此,以IgG格式测试了该抗体,该抗体随后能够从ORFeome噬菌体展示中复制结果,尽管原则上也检测到其他彼此不相关的结合区域。关于映射的MSR的最后也是最有趣的发现是关于GSM313-E9和GSM313-H8,它们是间接ELISA实验中唯一显示100%敏感性和特异性的抗体,并且是唯一结合这两个部分组成的序列的抗体第一和第二LD。
当使用单基因噬菌体展示时,可以减少分离片段的大小,从而减少用于ORFeome噬菌体展示的4种抗体的检测到的MSR(表3,另请参见文本S1和S2)。单基因噬菌体展示中的scFv-Fc抗体GSM130-H1和GSM133-A4的MSR分别小8.54%和48.70%。而IgG GSM133-E2的MSR大小降低了71.43%。对于GSM134-C1,只有单基因噬菌体展示会产生反应性序列,从而可以确定MSR。
讨论区
噬菌体展示主要用于产生抗体用于治疗和研究45。尽管如此,该技术最初是为表位检测而设想的46。因此,抗原噬菌体展示方法仍然很有效地找到用于诊断或疫苗开发的生物标记物47。本研究结合了三种不同的噬菌体展示技术。最初,抗体噬菌体展示用于从单核细胞增生李斯特菌的亚细胞级分中发现新的生物标记。还已经证明了针对这种复杂蛋白质混合物的抗体的产生已针对病毒,例如猪流行性腹泻病毒48和黄热病49和革兰氏阴性菌嗜肺军团菌50。但是,在病毒的情况下,靶标通常是已知的,与亚细胞级分相比,靶标的数量会减少。对于嗜肺乳杆菌,用于淘选的细胞预先用甲醛处理,这可以解释抗脂多糖(LPS)抗体的富集。一项类似的研究采用抗体噬菌体展示来产生针对单核细胞增生李斯特氏菌细胞的scFv 51。有趣的是,后一项研究采用了两种不同的淘选策略,所有阳性结果均会产生相同的抗体序列,从而产生相同的靶标。本研究证实了这一结果,因为执行了四种不同的淘选策略,其中三种导致针对同一靶标的有用抗体。尽管与先前发表的文章中描述的单个抗体相比,此处生成的四种初始抗体代表更高的数量,但是不同策略可以导致相同靶标的事实是进一步研究的有趣点。
事实上,大多数通过噬菌体展示对抗体产生的研究描述了使用纯化的抗原,这表明该方法可能是提供高品质的抗体更成功52,53,54,55,56。因此,在本研究中,利用这种策略产生了16种新抗体,其中四种显示出比最初针对细胞组分产生的四种最佳抗体更好的诊断性能(参见表3)。
噬菌体展示的另一个应用是展示整个ORFeomes和metagenomes。这种技术,它采用与从细菌基因组中,宏基因组或cDNA片段抗原建库中,描述了允许的使用血清或多克隆抗体的其他来源的寡肽新颖标志物的发现40,41,57,58。即使此ORFeome应用程序可用于本研究,使用抗李斯特菌的多克隆抗体,它将提供有关可用于间接检测的免疫原性蛋白质的信息。但是,由于目的是发展直接检测,因此使用抗体噬菌体展示代替ORFeome,以获得能够检测完整细胞中靶标的抗体。尽管如此,ORFeome被用于目标识别,否则通常可以通过基于MS的技术来实现。与IMS-MS方法相比,ORFeome噬菌体展示确定了相同的目标。在这种方法中,四种抗体中的三种显示出与IMS-MS相同的靶标(表S2),这表明ORFeome噬菌体展示可提供与IMS-MS类似的靶标识别信息,从而允许“仅噬菌体展示”方法(图 5)。
使用单基因噬菌体展示进行MSR分析后,大多数抗体的MSR长度为71至87个氨基酸,这是PDC-E2 LD之一的近似大小。这表明这些域的正确折叠对于所选抗体的识别是必不可少的,这一发现在以前的白喉毒素研究中也已注意到,其中C域的MSR具有≈150个氨基酸59。单基因淘选还显示GSM133-A4实际上识别了目标的两个LD,并使得确定GSM134-C1的MSR成为可能,而在ORFeome显示中没有显示结果。即使ORFeome也被报道可以在高分辨率的杂交瘤来源单克隆抗体中进行表位作图31,本研究的结果表明,单基因展示可以显着提高检测到的表位区域的分辨率。这证实了这样的事实,即使用类似方法针对白喉毒素定位的大多数抗体具有相对较短的MSR(14-38个氨基酸)59。
PDC是一种酶复合物,可催化丙酮酸转化为乙酰辅酶A,这是将糖酵解与柠檬酸循环(CAC)连接的步骤。由于其在细胞呼吸中的关键作用,该复合物在几乎每个生物体中均被保守,并由三种或四种不同的蛋白质组成60。尽管保持相同的功能,但复合物及其酶的结构在不同的生命形式中有所不同。在真核生物中,四种酶通常组成复合物,而原核细胞中不存在PDC-E3结合蛋白(PDC-E3BP)。然而,在原核生物中,复合物主要由三种酶组成,它们在革兰氏阴性菌和革兰氏阳性菌之间也显示出实质性的结构差异61。PDC结构之间的基本区别是构成组合系统核心的PDC-E2副本的数量。在真核生物中,提出了几种针对PDC的模型,它们可以包含40-60个PDC-E2副本以及12-20个PDC-E3BP。在革兰氏阴性菌中,核心由≈24个PDC-E2拷贝组成,而在革兰氏阳性菌中,核心也可以具有≈24个拷贝(形成八面体对称,立方体形状),或者更接近真核生物,≈ 60份(形成二十面体对称,十二面体形状)。考虑到主要针对PDC-E2的结构信息,以及本研究显示的间接ELISA结果,可以观察到一些相关性。本研究中使用的革兰氏阴性菌种与大多数测试抗体均无反应(只有GSM313-B12显示出交叉反应性),根据革兰氏阴性和革兰氏阳性之间已知的结构差异。此外,已知的结构粪肠球菌(本研究中使用的物种)和嗜热脂肪地芽孢杆菌呈二十面体对称。由于肠球菌是间接ELISA中显示大多数交叉反应的属之一,因此可能是李斯特菌属。PDC具有相同的对称性。
我们研究的一个令人费解的结果是在李斯特菌细胞外部检测到PDC 。已知在丙酮酸从胞质溶胶转运后,真核生物中的PDC在线粒体基质中起作用。但是,该复合物在基质中的确切位置尚未完全描述,因此,线粒体中与膜的任何接近性尚不清楚。同样,革兰氏阴性菌和革兰氏阳性菌中的PDC多见于细胞质中。然而,在细胞壁中也发现了一些其他代谢蛋白,例如1,6-果糖二磷酸二醛缩醛醛糖酶(FBA),主要是胞质的或附着在膜上的,因此可以在细胞表面上获得31,62。李斯特菌中的 PDC-E2它可能与FBA相似,是一种主要存在于细胞质中但通过膜到达细胞壁和细胞表面的蛋白质。在本研究中,使用细胞壁,膜和细胞质的淘选策略均能够提供针对PDC-E2的结合剂,从而证实了其在不同细胞部位的定位。进一步的研究可以解决细菌表面上出现的其他代谢蛋白是否为检测提供有用的生物标记,特别是考虑到李斯特菌表面根据环境进行了重塑63。
尽管PDC的代谢功能已明确定义,但该蛋白也可能与致病性有关。在结核分枝杆菌中,发现PDC-E2在具有免疫能力的感染人群中诱导强烈的细胞反应,也有助于细菌抵抗宿主反应性氮中间体64。在牛分枝杆菌中,PDC-E1β被鉴定为感染牛的一种免疫优势抗原,从而使得间接ELISA诊断测试的发展比商业化检测具有更好的性能65。肺炎分枝杆菌的PDC-E1β 具有纤连蛋白结合活性,被归类为月光蛋白66。在另一项研究中,沙门氏菌肠亚种的PDC-E1 。肠型血清型肠炎沙门氏菌被描述为一种毒力因子,一旦具有缺失或不具有基因的突变体显示出不太可能引起疾病的重要症状67。在缺乏PDC的金黄色葡萄球菌中也会出现诸如生长较慢和膜流动性增加等特征,表明酶68的结构影响。PDC-E2还提出了作为抗生素治疗的目标69,70和疫苗开发71对结核分枝杆菌。但是,到目前为止,尚无研究评估PDC的影响。李斯特菌属 在任何这些方面。由于其他代谢酶,如李斯特菌粘附蛋白(LAP),已显示出月光作用72,因此应进一步研究李斯特菌 PDC 的功能。
在间接ELISA中,某些李斯特菌属物种与大多数抗PDC-E2抗体的反应性降低,而某些非李斯特菌属物种则具有升高的反应性(图S4)。灰色李斯特菌,水生L. atictica和康奈尔李斯特菌在李斯特菌属物种中显示最低的反应水平,信噪比勉强高于1。尽管本研究中未研究的许多因素可能会导致反应性差异,但一些可能性和观察可能值得一提。灰李斯特菌在基因上被分类为一个独立的系统发育亚组(称为Murraya),表示可能影响蛋白质表达谱和细胞表面组成的特殊特征73。据此,当基于PDC-E2的序列分析系统发育时,灰色乳杆菌似乎也从该属的其他物种中分离出来,显示出最高的遗传距离(图S5)。水产李斯特菌与弗氏乳杆菌和弗洛里德乳杆菌属于另一个亚组(称为Mesolisteria)。然而,它与灰乳杆菌有一些共同特征,因为这两个物种都是已知的唯一通过葡萄糖代谢产生乙酰丁香的物种74。此外,与使用325种单拷贝基因进行分析的其他物种相比,这两个物种都显示出相似的遗传变化和最高的遗传距离73。当使用PDC-E2序列时,也观察到了类似的行为,其中灰色乳杆菌和水生乳杆菌都显示出最高的遗传距离(图S5)。至于康奈尔乳杆菌,它分为不同的亚组(称为Paenilisteria)。值得强调的是,本研究中该物种的最佳生长是在30°C时实现的,而不是大多数其他物种的37°C。奇怪的是,在这项研究中,所有在30°C下生长的物种都是该亚组的一部分,并以相同的系统发生簇出现73。此外,山茱L是唯一呈现低乳糖酸化的物种 74。至于目前尚未包括在本研究中的三种最新李斯特菌,预计抗PDC-E2的两种最佳抗体也可以很好地识别它们,因为它们的PDC-E2与靶标的身份相同与的李斯特菌是82.5%或更高(见表 S1)。
当分析非李斯特菌菌株的反应模式时,很明显芽孢杆菌属和肠球菌与某些抗体表现出升高的反应性。因此,许多研究描述了芽孢杆菌密切相关的李斯特菌,强烈表明表型的相似性75,76。尽管不经常在李斯特菌和肠球菌之间进行系统发育比较,但这些生物可能具有足够的相关性,可以共享一个表型,从而可以解释与某些抗体77的高反应性。此外,芽孢杆菌和芽孢杆菌的 PDC-E2肠球菌显示出与单核细胞增生李斯特菌相当的身份(> 50%),这可能解释了交叉反应性。仅革兰氏阳性菌种显示出这种反应的事实表明,PDC-E2暴露于细胞表面的可能性更高,这可能是PDC 61结构相似的结果。总之,本研究中显示的抗体对PDC-E2的识别可能取决于多种因素,例如靶标的细胞位置(表型),与李斯特菌 PDC-E2的同一性以及复合物的整体结构。
有趣的是,认识到PDC-E2的两个LD对诊断性能无益。预期在相同分子中识别两个LD将导致更高的信号,这可能导致更高的灵敏度。尽管如此,尽管GSM313-F7显示出比大多数抗体更强的反应,但是反应性的提高似乎并不是识别LD且不有助于敏感性的抗体的趋势(图S6)。另外,两个LD的识别可能与特异性降低有关,因为具有这种识别模式的4个scFv-Fc中有2个显示与非李斯特菌的反应增强种类和AUC <0.8(对于GSM313-F7为0.7895,对于GSM313-G10为0.6632),将它们置于诊断性能的最后五个位置(请参阅表2;其他两种抗体是GSM313-D10和GSM133-A4,分别显示AUC = 0.9789和0.8947)。
具有最佳诊断性能的两个scFv-Fc(GSM313-E9和GSM313-H8)显示出有趣的模式,因为它们识别出第一个LD和由PDC-E2的两个LD融合而形成的合成LD,可能是库构建过程中随机碎片和连接的结果。另一个有趣的抗体是GSM130-H1,因为它是最初产生的针对蛋白质级分的性能最好的抗体。值得一提的是,该抗体是使用李斯特氏菌从淘选策略中选择的细胞壁蛋白,这表明其更好的性能可能与PDC-E2在该亚细胞部分所呈现的特定构象有关。GSM133-E2的情况也值得一提,因为它特定于PDC-E2,但存在多个结合位点,可能例示了多特异性的情况。多特异性抗体是公知的识别多于一个表位具有高度特异性78,79,80。从LD区域外部检测到的肽的疏水成分仅稍高(氨基酸的45-55%,而LD区域为≈40%),但考虑到经常造成非特异性反应的高疏水性肽通常含有较高的疏水性肽> 50%的疏水氨基酸和<25%的酸性成分81。这样,GSM133-E2可能不是本质上非特异性的,而是表现出降低诊断性能的多特异性。如果是这种情况,则该抗体可以结合同一靶标的不同表位,而不是不同靶标中的相似表位。
此处描述的体外技术(抗体展示,ORFeome展示和单基因展示,请参见图 5)的组合可用于鉴定新型生物标志物(PDC-E2),产生可用于检测李斯特菌的单克隆抗体。(GSM313-E9和GSM313-H8)中的蛋白质,并表征潜在的抗体-抗原相互作用。这样,用于检测该属的有用靶标和生物分子的列表增加了。在商业适用的测试中,有必要进行进一步的研究以评估这些分子。
方法
细菌培养和李斯特菌蛋白分离
来自李斯特菌属的细胞壁,膜和细胞质的蛋白质。如82所述制备细胞。ELISA用于确定蛋白In1A(存在于细胞壁中)和FBA(主要存在于细胞质和膜中)的存在。为此,将细胞壁,细胞质和膜级分分别在1:100、1:100和1:400的磷酸盐缓冲盐水(PBS)中稀释,并包被在ELISA Costar板(Corning)上。将板用2%(w / v)奶粉稀释,该粉在PBS中稀释,补充0.05%(v / v)Tween-20(2%MPBS-T),并与单克隆抗体2D12(小鼠IgG抗InlA,1 µg / mL)22和3F8(小鼠IgM抗FBA,1 µg / mL)31。最后,将板与针对2D12的抗小鼠IgG Fc特异性抗体(Sigma)或针对HF共轭的针对3F8的抗小鼠IgA,G,M(Antibodies Online)进行孵育。用TMB溶液(TMB-A:50 mM柠檬酸,30 mM柠檬酸钾,pH 4.1; TMB-B:90%(v / v)乙醇,10%(v / v)丙酮; 10 mM四甲基联苯胺进行反应; 1 mL 30%H 2 O 2;将19份TMB-A与1份TMB-B混合,用1 NH 2 SO 4终止,并以620 nm为参考在450 nm处分析板。
李斯特菌蛋白级分或rPDC-E2的抗体淘选以及单克隆scFv筛选
在李斯特菌蛋白组分在不同的淘选策略使用。总体而言,分别使用人类纯抗体噬菌体展示文库HAL9和HAL10 83进行了四种方法。所有这些策略均始于将Hyperphage(Progen)包装的文库与淘选区溶液(1%奶粉,1%BSA稀释于PBS-T中),无毒李斯特菌 DSM 20649细胞预孵育到三个ELISA孔中壁或热灭活的枯草芽孢杆菌 168 NCIB10106。热灭活的枯草芽孢杆菌细胞通过在BHI中于37°C,250 RPM过夜生长而制备。收获细胞(2,600× g,7分钟,4°C),悬浮在碳酸盐-碳酸氢盐缓冲液(75 mM Na中)中2 CO 3,75mM的碳酸氢钠3,pH值9.7),直到OD 600 = 1.0(≈10 9个 细胞/ mL),在90℃下加热10分钟,并储存于-直到使用20℃。进行了以下淘选策略:(1)单核细胞增生李斯特氏菌 ATCC 7644细胞壁级分,在枯草芽孢杆菌(1×)和PanningBlock(2×)上预孵育;(2)单核细胞增生李斯特氏菌 ATCC 7644细胞质级分,且预孵育相同;(3)单核细胞增生李斯特氏菌 ATCC 7644膜级分,具有相同的预孵育;(4)相同的策略“1”,但对预孵育无害利斯特氏菌 DSM 20649的细胞壁。
为了筛选,如上所述涂覆细胞壁,膜和细胞质级分。针对蛋白质级分,单核细胞增生李斯特氏菌 ATCC 7644,无毒李斯特菌 DSM 20649或枯草芽孢杆菌 168 NCIB 10106对产生的单克隆scFv进行了阴性对照测试。其余程序按先前发布的步骤进行84。
进行了针对重组PDC-E2的淘选的类似程序。但是,这一次,将文库HAL9和HAL10在一个单独的淘选孔中混合,用1 µg PBS稀释的蛋白质包被。为了筛选,使用在PBS中稀释了200ng /孔的重组蛋白的板代替蛋白级分。
scFv-Fc和IgG的克隆和生产
来自上一步骤的针对蛋白质级分的选定scFv将其基因亚克隆到带有小鼠IgG2a Fc的pCSE2.6载体中,用于生产scFv-Fc。在GSM133-E2的情况下,也使用载体pCSEH和pCSL 85将其作为人IgG1产生。如先前所述86进行HEK293细胞的克隆和生产。纯化后,每种scFv-Fc抗体在李斯特菌上的结合。与scFv筛选相似,通过ELISA检查细胞。这次,使用√10倍系列稀释抗体。
具有蛋白质级分的免疫印迹,以及质谱鉴定目标
通过12%SDS-PAGE分离来自无毒李斯特菌 DSM 20649 细胞壁和细胞质的蛋白质部分,然后转移至甲醇活化的PVDF 0.45 µm膜(Roth)。将膜用2%MPBS-T封闭过夜,然后在室温下分别与1 µg / mL的scFv-Fc一起孵育1小时。山羊抗小鼠IgG Fc特异性HRP共轭抗体(1:40,000; Sigma)被用作二抗,DAB溶液(6 mg 3.3-二氨基联苯胺四盐酸盐; 10μL30%H 2 O 2 ; 9 mL PBS; 1 最后加入15mL NiSO 4 250mM溶液。
用100 µL SureBeads蛋白A磁珠(Bio-Rad)进行免疫磁分离(IMS),并用10 µg每种抗体包被10分钟。将无病菌DSM 20649的细胞质制剂在PBS中以200:L的比例稀释1:2,并与磁珠孵育1小时。随后,用20 mM,pH 2.0的甘氨酸缓冲液洗脱5分钟。然后,添加10%(v / v)的Tris-NaCl 1M,pH 7.4。在SDS-PAGE中分析洗脱样品,从中切除涉及目标的蛋白质以进行质谱(MS)分析。使用与IMS相同的scFv-Fc作为一抗,以与上述相同的方式进行相应的免疫印迹。
如先前所述87处理从SDS-PAGE凝胶中获得的条带。然后将纯化的肽直接洗脱,并与α-氰基-4-羟基肉桂酸共结晶到锚定芯片靶标(Bruker)上,进行基质辅助激光解吸电离(MALDI)。MS分析是使用Ultraflex飞行时间(TOF)和TOF质谱仪(Bruker)手动进行的。用肽质量指纹图谱方法鉴定肽。使用Bruker's Biotools和室内Mascot服务器(Matrix Science v。2.4.1)进行MS数据处理。数据库搜索仅限于李斯特菌属 国家生物技术信息中心的数据库(NCBI,版本20170811)和蛋氨酸氧化被认为是一个变量,也被固定为碘乙酰胺修饰。
或女性展示
从单核细胞增生李斯特氏菌 ATCC 7644的随机基因组片段构建的噬菌体文库用于对获得的抗体(GSM130-H1,GSM133-A4,GSM133-E2和GSM134-C1)进行淘选,以鉴定抗体的靶标。先前的研究31中描述了使用的库。简而言之,将1μg抗体包被在ELISA板上。对于库的预先清除,2×10 11 将CFU在其他三个不同的孔中于室温孵育20分钟:(1)PanningBlock溶液;(2)不相关的scFv-Fc;(3)溶液中不相关的scFv-Fc加10μg的scFv-Fc。其余步骤如前所述进行,只是只进行了一次淘选。通过ELISA鉴定反应性克隆后,对其进行测序并用BLASTn进行分析。
为了确认和验证筛选ELISA的结果,使用先前描述的程序43,以较大规模产生了阳性命中的单克隆噬菌体。在间接ELISA中滴定含有产生的单克隆噬菌体的最终悬浮液,以确认与每种抗体的结合。为此,将噬菌体包被到ELISA孔中,从10 9 CFU / 孔开始将它们稀释12倍√10倍。将针对各个单克隆噬菌体的抗体调整为2 µg / mL,并与山羊抗小鼠IgG Fc特异性HRP偶联抗体(1:40,000; Sigma)一起孵育1小时。
李斯特菌细胞显微镜
单核细胞增生李斯特氏菌菌株。4b DSM 15675在BHI中生长(16小时; 37°C; 110 RPM)。然后离心2 mL(2,500× g; 2分钟),用PBS洗涤2次,并与三种不同的抗体孵育:(1)抗内在蛋白A scFv-Fc作为阳性对照;(2)以无关的scFv-Fc为阴性对照。或(3)GSM133-A4 scFv-Fc(抗PDC-E2,作为实验分子)。将这些抗体稀释在2%MPBS-T(终浓度5 µg / mL)中,并在室温下孵育1小时。将细胞用PBS-T洗涤3次,并在室温下与抗小鼠IgG Alexa 647二抗(Thermo Scientific)在室温下孵育1小时,该抗抗体在2%MPBS-T中以1:500稀释。用相同的方法洗涤细胞,并将其悬浮在含有封固剂的相应体积的DAPCO(1,4-二氮杂双环[2.2.2]辛烷)中。如前所述进行样品制备88。使用带有Cy5(650 / 13–705 / 72)滤镜的Nikon Eclipse Ti倒置显微镜进行成像。使用Nikon N Plan Apochromat lλ×100 / 1.45油物镜和ORCA FLASH 4.0 HAMMATSU相机拍摄荧光z堆栈和相差图像。使用NIS元素成像软件V4.3(Nikon)以及3D Landweber Deconvolution算法对图像进行处理。
重组靶标PDC-E2的克隆,在大肠杆菌中表达及纯化
PDC-E2的基因(代码:AL596167.1:98629-100260)和蛋白质(代码:WP_010990728.1)序列从GenBank获得,并用于设计用于亚克隆的引物。含有限制性酶切位点Nde I和Not I的引物按如下顺序排列:正向5'-AATTC CATATG GCATATTCATTTAAATTACCGGATATCG-3',和反向5'-ATTG CGGCCGCC ACCTCCATTAGTAATAATTCTG-3'。按照生产商的说明,使用Phusion高保真DNA聚合酶(Thermo Scientific),使用单核细胞增生李斯特菌 ATCC 7644的基因组DNA进行PCR。扩增的基因亚克隆到pET21a(+)(Novagen)中。
为了表达,将含有pET21a(+)/ PDC-E2的大肠杆菌 BLR(DE3)在10 mL LB-A(Luria-Bertani含有100 µg / mL氨苄青霉素)中于37°C,250 RPM下培养16 h。然后,接种200 mL LB-A直至OD 600 = 0.1,并在相同条件下生长直至OD 600 = 0.6-0.8,然后用IPTG诱导至终浓度125 µM,并孵育4小时。通过离心(10,000× g; 10分钟; 4°C)收集细胞,将其悬浮于20 mL结合缓冲液(5 mM咪唑,0.5 M NaCl,20 mM Tris-HCl,pH 7.9)中,并进行超声处理(3×2脉冲周期的最小值)。将悬浮液离心(16,000× g; 10分钟;4°C),并将上清液转移至新试管中。用抗6xHis抗体(Dianova)进行电泳和免疫印迹以确认表达。最后,使用洗涤液(60 mM咪唑,0.5 M NaCl,20 mM Tris-HCl,pH 7.9)和洗脱缓冲液(0.5 M咪唑,0.5)在重力流柱(GE Healthcare)中进行Ni 2+亲和纯化。 NaCl,20 mM Tris–HCl,pH 7.9)。如先前发表的[ 89]所示,用PBS稀释的洗脱液对蛋白质进行透析,以逐渐降低NaCl的浓度。
李斯特菌属的间接ELISA 。检测
使用的菌株(表4和5)获自Leibniz Institute DSMZ-德国微生物和细胞培养物保藏中心。从具有BHI的冻干原液中回收每种细菌,使其在10 mL BHI中生长,并划线到BHI-琼脂平板上。例外的是副干酪乳杆菌,它总是在MRS或MRS-琼脂中生长。根据其处理说明,细菌分别在30或37°C,80或110 RPM摇动下生长。必要时,通过使用厌氧蜡烛罐创建微需氧条件。
对于ELISA,从板中取出每个菌株的单个菌落,并在标准条件下在10mL的相应培养基中生长(表4和5)。然后,通过离心收集所有细胞,用PBS洗涤两次,并悬浮在碳酸盐-碳酸氢盐缓冲液中至OD 600 = 1.0。ELISA板的制备是在4°C下用100 µL /孔的细胞悬液包被过夜。将板在室温下用2%MPBS-T封闭1小时,然后在室温下用在3%不同浓度的2%MPBS-T中稀释的不同抗体进一步孵育1小时:(a)估计的EC 50(“ EC 50 ” ); (b)在EC 50上方√10稀释(“ EC 50+”);(c)在EC 50以下(“ EC 50- ”)稀释√10 。作为二抗,使用缀合有山羊抗小鼠IgG Fc特异性HRP(1:30,000; Jackson ImmunoResearch Laboratories)。具有非相关性scFv-Fc的孔,以10 µL / mL的比例稀释√10倍,仅含有二抗的孔作为对照。
根据每个读数计算信噪比,然后除以每个板的二抗对照中三个孔的平均值。抗体浓度和细菌菌株的每种组合均在单个孔中进行,并在独立的平板中重复两次,尽管只选择了一个平板进行统计分析。将结果值用于使用GraphPad软件(Prism,v 5.01)的接收器工作特性(ROC)分析中,其中计算了灵敏度和特异性以及置信区间(CI)。为了便于描述结果,曲线下面积(AUC)值也用于表示此数据。
PDC-E2的单基因噬菌体展示
单基因文库与所述基因编码PDC-E2内置,如前所述43,44。淘选与用于靶标发现的ORFeome淘选相似地进行,其中包被1μg的每种抗体。每个淘选井 使用10 9 CFU,仅进行第一轮淘选。如上所述进行筛选,并将反应性克隆送去测序。将所得序列与ClustalOmega软件进行比对,以定义最小识别序列(MSR),该序列定义为每个选定克隆上存在的最短序列。
PDC-E2的序列分析
通过MS和ORFeome噬菌体展示检测到的目标PDC-E2的蛋白质序列用于BLASTp中,以查找间接ELISA中每个物种的同源物。使用PDC-E2序列(GenBank代码:WP_107899613.1)作为查询,使用在线成对比对工具EMBOSS Needle 90(表S2)计算同一性和相似性。使用SMART在线软件91分析了同一序列,该软件允许定义其不同区域。
|
|