摘要
背景
全基因组关联研究(gwas)已经确定了很多非编码生殖系单核苷酸多态性(snp)与患癌症的风险增加有关。 然而,如何将这些单核苷酸多态性影响患癌症的风险在很大程度上仍然是未知数。
方法
我们用系统生物学的方法分析风险的监管作用snp在13个组织。 通过使用数据从Genotype-Tissue表达式(GTEx)项目,我们进行了一个表达式数量性状位点(eQTL)分析。 我们代表意义顺式和反式-eQTLs边缘在组织eQTL由两部分构成的网络。
结果
每个组织eQTL网络社区组织了一套单核苷酸多态性和功能相关的基因。 癌症snp的映射到这些网络时,我们发现,在每一个组织,这些单核苷酸多态性明显的过多社区丰富的免疫应答过程,以及组织功能。 此外,癌症snp的更可能是“核心”的社区,影响许多基因的表达在相同的生物过程。 最后,癌症snp的优先目标癌基因和肿瘤抑制基因,这表明他们可能改变这些关键的癌症基因的表达。
结论
这种方法提供了一种新的方式来了解基因对癌症风险的影响,提供了一种生物上下文解释GWAS癌症研究的结果。
背景
癌症往往由于体细胞突变致癌基因和肿瘤抑制,经常出现由于环境因素如紫外线、烟草、烟雾或致癌的化学物质。1,2,3.遗传性癌症代表5至10%的癌症和的特点是家庭病史,发病和更高的比平常年轻时代主要在多个器官癌症的可能性。 他们经常与生殖系改变癌基因和肿瘤抑制基因。4但除了这些明显的癌症“司机”,人们普遍认可,其他遗传因素在癌症发展和发展中发挥作用。 全基因组关联研究(gwas)已经确定生殖系单核苷酸多态性(snp)与改变癌症风险相关(“癌症snp的”)。 然而,许多单核苷酸多态性确定通过gwas落入non-genic地区,很难解释他们在疾病发展的生物作用,发展和对治疗的反应。5
人口生殖系癌症风险SNP的频率通常是反关联效应,计算之间的相对风险携带突变的人,那些不。6虽然少数罕见变异的功能强劲的影响研究,对函数的常见风险变异影响小,出席中频一般人群。 在snp在GWAS通过全基因组意义的目录栏与风险升高的一个或多个癌症,大多数有优势比< 1.3,大部分不属于基因(位于“non-genic”地区),这表明他们可能在基因表达的调控中发挥作用。6,7
表达数量性状位点(eQTL)分析测试协会之间的SNP位点的基因型和基因表达水平,和一个eQTL协会可以提供证据,SNP的监管作用。 多项研究表明,单核苷酸多态性与特征和疾病相关的GWAS eQTLs富集,从而加强他们可能发挥监管作用的假设。8,9很多研究也发现,癌症snp基因表达水平的影响。10,11,12,13然而,大多数生殖系癌症snp没有生理上的特征,及其总体的未知功能的影响。
这种差距在我们理解的癌症snp可能是由于其固有的特点。 除了小影响宏观表型(癌症),癌症snp也通常有小影响单个基因的表达。14此外,由于许多基因展览组织表达,很难描述癌症snp的监管作用靶基因不表达最经常组织学习,如全血。14,15最后,由于健康的细胞转变成癌细胞与许多基因组和转录组变化有关,我们不能用肿瘤细胞的研究调查的影响监管风险snp pre-tumour细胞。
新的数据集可以帮助阐明癌症snp的角色。 大规模研究,如Genotype-Tissue表达式(GTEx)项目,提供基因组和转录组数据从数以百计的个人和几十个十几组织,16从而使癌症的影响单核苷酸多态性在多种组织进行评估,包括那些在最相关的影响。
在这项研究中,我们使用一个系统生物学的方法来描述生殖系癌症风险的监管作用snp在13个不同的组织(补充表1通过使用数据从GTEx项目v6.0)。 在每一个组织,我们执行一个eQTL分析和代表独联体- - -反式-eQTLs通过使用由两部分构成的网络。 然后映射生殖系癌症SNPs和癌基因和肿瘤抑制基因网络和使用网络的属性来识别癌症单核苷酸多态性的影响的生物功能和途径。
我们发现尽管癌症单核苷酸多态性的分布在网络,它们浓缩在一个小的社区与免疫反应和识别病原体,强调在癌症免疫过程的重要性。 特别是,癌症snp优先映射到社区丰富的基因属于主要组织相容性复合体(MHC),表明潜在的更大的作用在癌症免疫过程比预期的风险。 我们还发现癌症snp的所占比例在当地社区中心(“核心snp”),通过调节多种基因参与同一生物功能独联体而在反式。 最后,我们发现癌症snp的优先位于癌基因和肿瘤抑制基因的启动子,并更有可能比预期的机会影响这些癌症相关基因的表达水平。 这个分析演示了使用组织两偶eQTL网络的力量作为一个框架来研究生殖系单核苷酸多态性可以协调放松生物功能的表达,可以导致癌症的风险增加。
方法
GTEx数据预处理、过滤和合并
我们下载NHGRI GTEx v6.0估算基因分型数据和RNA-seq数据(phs000424.v6。 从dbGaP p1, 2015-10-05版)(批准协议# 9112)。 RNA-Seq数据预处理是通过使用Bioconductor R纱包17和正常化tissue-aware方式通过使用光滑的分位数正常化Bioconductor R qsmooth包。18我们识别和移除GTEx-11ILO misannotation由于潜在的性。 我们也过滤掉性染色体和线粒体基因,保留29242个基因。 我们排除了五个性别组织(前列腺、睾丸、子宫、阴道、卵巢)和分组的小腿的皮肤样本(太阳暴露)和耻骨弓上的地区(太阳未曝光)基于整体基因表达这些网站之间的相似性。 对于我们的分析,我们只考虑组织我们都RNA-seq基础估算数据至少200人。 十三组织符合所有标准在预处理和被用于后续的分析(补充表1).
GTEx RNA-seq和基因分型数据映射的财团GENCODE版本19日是基于人类基因组构建GRCh37。 p13(2015年9月)。 RNA-Seq我们进行主成分分析数据在每一个组织,并寻找潜在的混杂元数据元素通过寻找那些与前十RNA-Seq主要组件。 对于所有组织,我们占的地方捐赠招募,RNA提取设备的影响,提取RNA的质量,死亡,死亡和组织开始之间的时间间隔采样和捐赠者是否在通风机立即死亡之前使用R limma包。19
eQTL映射和双方的网络建设
eQTL分析,我们排除了snp分析如果他们有一个叫率在0.9或轻微的等位基因频率在任何组织< 5%。 如果样品中基因表达被认为是其阅读数大于或等于6。 基因表达在不到10的样本组织被eQTL分析的组织。 纠正不同程度的掺合料的非裔美国人的主题,我们使用前三个主成分的基因分型GTEx协会提供的数据,包括这些在我们eQTL模型。
我们用R MatrixEQTL包20.计算eQTLs添加剂的线性模型,包括年龄、性别和种族背景,以及前三个基因型电脑不:
我们检测和单核苷酸多态性基因表达水平之间的联系独联体和反式,我们定义独联体在1 MB的单核苷酸多态性与基因的转录起始站点基于映射通过Bioconductor R biomaRt包。21 P值调整为多个测试通过使用Benjamini-Hochberg修正独联体- - -反式-eQTLs分开,只有那些有调整P值< 0.2被用于后续的分析。
社区识别
对于每一个组织,我们代表了重大eQTLs作为双方的网络的边缘节点连接单核苷酸多态性和基因。 对于每一个网络,我们我们的分析集中于giant-connected组件,包含成千上万的基因和成千上万的snp。 其他连接组件被排除在分析由于其小尺寸(每一个都包含< 50基因和不超过2社区)。 确定高度连接社区eQTL单核苷酸多态性和基因的网络,我们用R秃鹫包,22它最大化的一式两份的模块化。23递归的集群识别和优化可以计算慢,我们计算一个初始群落结构赋值加权,gene-space投影,通过使用一个快速unipartite模块化最大化算法24在R igraph包,25然后迭代地聚集在一个社区结构对应于最大双边的模块化。
一式两份的模块化定义在情商。1),\ \(米)在网络链接的数量,\ ({\ widetilde{一}}_ {ij} \)是右上角块网络的邻接矩阵(二进制矩阵1代表一个SNP和基因之间的联系和0),\ ({k} _{我}\)SNP的程度吗我\ \ (),j d \ ({} _ {} \)基因的程度吗\ (j \)和我\ (C {} _ {} \),j C \ ({} _ {} \)SNP的社会指标我\ \ ()和基因\ (j \),分别。
风险单核苷酸多态性
我们下载了NHGRI-EBI GWAS目录(2017年4月24日,访问版本v1.0)从EBI网站(https://www.ebi.ac.uk/gwas)。 我们过滤协会P值>\ (5 \ * 1 {0}^ {8}\)并提取单核苷酸多态性与患癌症的风险。 我们剩下的snp映射到GTEx数据。 具体来说,我们决定LD阻塞使用plink2——选择最多5 MB的块大小26和其他选项设置为默认值,这意味着两个snp被认为在LD的底部90% d '置信区间> 0.70,和置信区间的顶部至少0.98。 我们认为所有snp在同一个LD块全基因组重要癌症snp。
癌症基因
我们使用来自两个数据库的信息,网络的癌症基因27和宇宙的人口普查,28创建一个列表一般的基因突变在癌症,癌症基因(补充表年代2),包括癌基因和肿瘤抑制基因。 我们这些癌症基因映射到GTEx eQTL网络。
我们测试了癌症snp是否优先位于癌症基因的启动子。 我们下载转录开始站点(TSS)位置对所有基因存在于GTEx运用数据库的数据29,30.并定义了发起人的−750 / + 250 - bp每个TSS周围地区。 我们使用确切概率法确定癌症基因启动子在癌症snp的丰富。 我们使用LD块而不是单核苷酸多态性分析,对连锁不平衡。
我们还测试了癌症snp是否比预期更频繁地与癌症有关的基因。 在每个网络,我们计算每个SNP的“癌症学位”通过计算重要的癌症基因的数量与每个SNP基于我们eQTL关联分析。 我们比较了癌症风险之间分布程度和non-cancer-risk snp考虑全球学位分配使用\ (1 {0}^ {6}\)重采样。 我们使用了Mann-Whitney U检验和比较U真正的和重新取样数据之间的值。
识别eQTL社区丰富的癌症snp
我们测试了eQTL社区进行浓缩的癌症snp使用确切概率法。 我们定义包含癌症snp的癌症LD阻塞的。 在每个网络,每一个癌症,我们测试是否个人社区风险单核苷酸多态性丰富,通过整个网络作为背景。 考虑一个社区作为癌症单核苷酸多态性丰富,我们使用一个阈值最低的四块LD社区。
SNP核心分数计算
我们定义了一个SNP eQTL网络核心得分的SNP对模块化的网络社区的贡献。 为国民党我\ \ ()在社区\ \ (h),其核心的分数,\ ({Q} _ {ih} \),被定义为情商。2)。 正常化snp在社区,我们在下游的社区成员占测试(方程式。 (3.)和(4)),更好的占社会变化与正常化方法用于参考。22
基因本体功能类别浓缩
我们的列表中提取基因在每个社区在每个组织网络,并使用R GOstat包31执行tissue-by-tissue分析群体中基因本体生物过程在每个社区丰富癌症snp。 我们参考集包括所有在场的基因在相应的组织网络。 社区被认为如果FDR-adjusted明显浓缩在一个特定的类别P价值是\ \ < 0.05 (\)。
癌症SNP的核心分分析
我们比较SNP核心之间的分数分布的癌症相关的SNP fr和那些没有与性状或疾病相关的组织网络利用似然比检验(轻轨车)。 在我们的设置中,轻轨交通评估线性模型,其中包括癌症风险状态(是否情商。(4))符合观测数据比线性模型不包括这个变量(Eq。3.))。 SNP核心分数的分布(\ ({Q} _ {ih} \))不是统一的社区,我们增加了社区的身份作为协变量的线性回归。 在方程式。 (3.)和(4),\ ({Q} _ {ih} \)SNP的核心分数吗我\ \ ()在社区\ \ (h),\ (n \)社区组织的数量。我(GWAS = 1) \ \ ()是一个指标函数等于1如果SNP与更高的风险在GWAS患上癌症,等于0如果不与任何性状或疾病相关。 单核苷酸多态性与性状或疾病相关的风险患上癌症被过滤掉。k C 是一个指标函数等于1如果苏格兰民族党属于社区\ (k \)否则,等于0。
控制单核苷酸多态性之间的连锁不平衡,我们提取的中值\ ({Q} _ {ih} \)癌症SNPs和non-GWAS snp对每个LD块,并使用这些值作为输入的线性回归。
结果
癌症snp位于非编码区域
我们定义一组的癌症snp基于NHGRI-EBI GWAS目录(加入日期:2017-04-24); 我们提取一组872个snp从565年独立的连锁不平衡(LD)块(在全基因组关联意义le 5 \ \ (p \ * 1 {0} ^ {8} \))与135独特的特征和疾病与癌症相关,代表41癌症类型(见补充表3.)。 大多数的癌症snp与只有一个相关的癌症类型; 只有6%是与两个或两个以上的癌症,而只有2%有超过三个癌症。 相比之下,大多数癌症类型(82%)与多个独立的单核苷酸多态性有关,与相关的独立的单核苷酸多态性的数量介于1 (b细胞非霍奇金淋巴瘤,心脏胃癌,慢性粒细胞白血病,脑膜瘤,non-melanoma皮肤癌,小肠神经内分泌肿瘤和零星的垂体腺瘤)和95年(前列腺癌)。
研究癌症的基因组位置单核苷酸多态性时,我们发现他们的个人影响患癌症的风险也通常小超过99%的癌症snp下有优势比3。 作为其他特征和疾病的观察,32我们发现只有9.7%的癌症snp其实或剪接变体SNPs, 40%是intronic,其余的被标注为“监管变体”或“基因间。 缺乏明确的已知生物功能的基于SNP位置表明许多剩余的91.3%可能发挥监管作用。 支持这种潜在的监管作用,我们发现,3.3%的癌症snp基因启动子中定义为−下降750 / + 250个基点在转录起始站点(TSS),而只有0.9%的non-cancer-risk snp位于启动子(重采样P价值le 1 \ (p \ {0} ^ {6} \))。 此外,癌症snp通常位于附近的基因TSS其中17.0%在5 kb TSS和84.0%的下降在100 kb,而只有8.0%和25.4%的non-cancer-risk snp位于这些区域,分别(重采样P值le 1 \ (p \ {0} ^ {6} \)).
癌症snp调节癌症相关的生物功能
生物功能的描述这大量的小的影响,基于监管、癌症SNPs,我们进行了系统的基因型和RNA-Seq eQTL分析通过使用来自GTEx v6.0的数据。 经过过滤和正常化GTEx数据,消除组织有不到200人的样本,我们只剩下13个组织基因表达和基因型数据(12个主要组织和1细胞系,看到补充表1)。 我们使用MatrixeQTL,20.纠正报道的性别、年龄、种族背景和三大基因型主成分,计算eQTLs独联体和反式在±1 - Mb窗口周围的基因(参见“方法”部分)。 我们使用相同的GTEx基因表达数据和过滤步骤在我们之前的研究中,33但修正四个潜在的混杂因素,已经被证明可以稍微影响转录组简介:提取RNA的质量,死亡的地方(在事故现场,救护车转移期间,医院,等等),死亡和组织开始之间的时间间隔采样和捐赠者是否之前在通风机立即死亡。 尽管有这些差异,eQTL结果很大程度上获得以前(斯皮尔曼相关ρ\ (\ \)从0.99到1时,通过使用eQTLβ-values分析计算,并计算通过使用时从0.89到0.94P值),结论与前面的论文都是复制。
对于每个十三的组织,我们代表的是重要的独联体- - -反式-eQTLs作为由两部分构成的网络,节点或单核苷酸多态性基因和边缘单核苷酸多态性和基因之间的联系非常重要。22,33增加的规模最大的连接组件,因为网络中心措施更敏感比边缘假阳性,假阴性34,35我们放松罗斯福截止,包括所有与罗斯福eQTLs q值低于0.2。 我们获得了13个组织网络包含57641年至431036年(ATA-aorta)和(THY-thyroid)单核苷酸多态性(所有13个组织中值= 198226),对应于3550年和34016年之间LD块(值= 15514),1090年和10003年之间基因(值= 4820)。
我们用R秃鹫包22的13 eQTL网络识别社区,定义为一组单核苷酸多态性和基因相互紧密连接比预计将偶然(见部分)的方法。 一式两份的模块化措施是否在社区网络的结构基因和单核苷酸多态性更容易与他们社区的其他成员比其余的网络,从0.83到0.97(值= 0.95)。 它表明,这些网络是高度模块化的,单核苷酸多态性和基因分组定义良好的社区。 的13个组织,我们发现29至177(值= 124)的社区。 然后我们通过测试群体中基因功能注释这些社区带注释的基因本体论(去)生物过程36(补充表4)。 我们发现一些社区丰富的基因在生物功能共享整个十三组织(免疫、基因表达调控和rna代谢),而其他组织(如心脏左心室心肌收缩和平滑肌收缩在食管肌层,这是一个平滑的肌肉)。 基因本体论浓缩和网络模块化类似Fagny et al。33
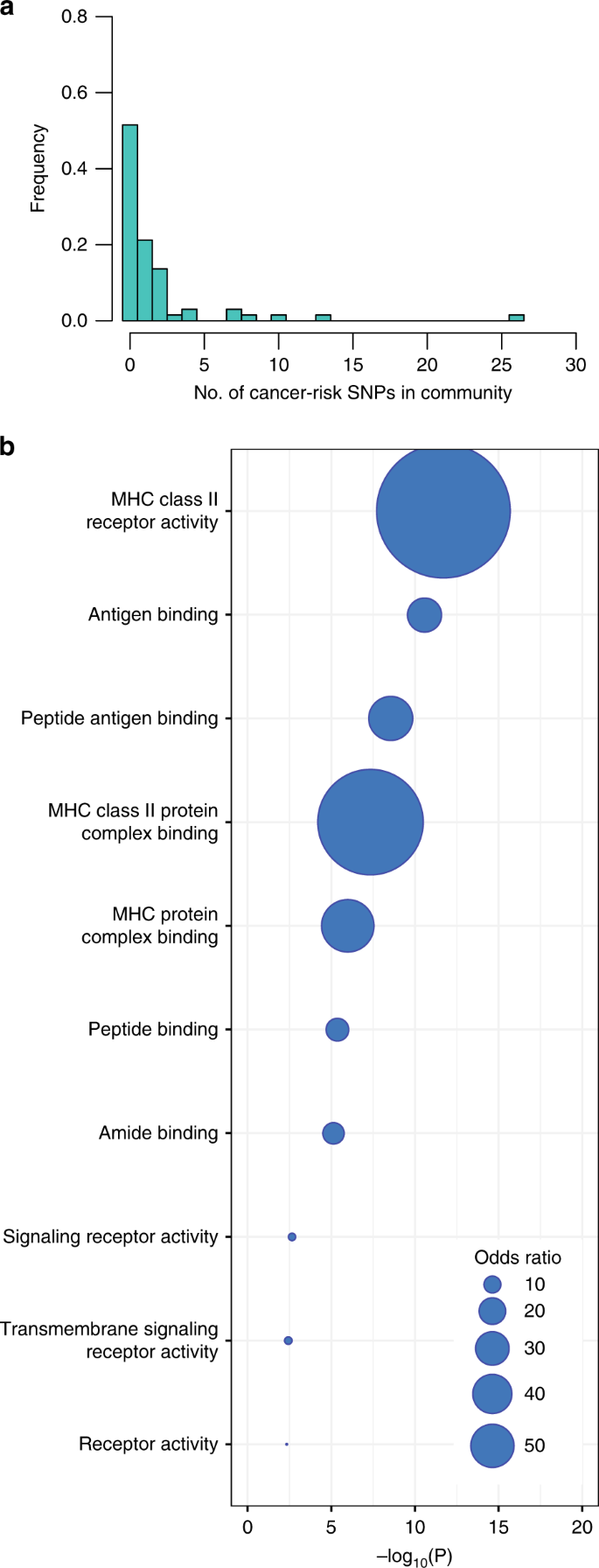
我们绘制了癌症snp eQTL网络为每个13组织。 872年的癌症SNPs, 582人一个eQTL或强大的连锁不平衡(r \ ({} ^ {2} \ > \ 0.8 \),见方法)eQTL至少1中至少一个基因的13个组织,确认这些snp的监管作用。 在13 9组织,这些癌症eQTLs略的丰富反式-eQTLs相比non-cancer-risk eQTLs,优势比从0.91在心脏左心室(费舍尔测试\ (p = 1.00 \)甲状腺(7.84)P= 9.51×10−49,补充表5)。 其中582癌症SNPs, 512映射到网络giant-connected组件(直接或通过加入一个强大的LD块)至少在一个组织。 这些snp映射到社区与广泛的生物学过程相关。 根据组织,21%(心脏左心室)和49%(肺)的社区包含至少一个癌症SNP。 然而,大多数社区只包含一个或两个癌症snp(表1和无花果。1a)。癌症snp的完整列表映射到每个13中的社区组织和相应的基因本体生物过程中提供了补充表6。 这些结果提供了一个可搜索的版本http://networkmedicine.org: 3838 / cancer_eqtl /。
然后我们测试每个组织中的每个社区浓缩的癌症snp。 因为研究表明,GWAS点击率最高并不总是因果SNP,最强,他们通常不对应eQTL冲击,我们包括所有单核苷酸多态性与癌症SNP的LD富集分析。 我们从相同的LD块和汇集snp注释作为癌症LD的块或不是癌症LD的街区。 我们这些LD块用于浓缩试验。 通过使用确切概率法,我们发现2 - 8(值= 4)在每个组织,丰富社区癌症snp(汇集所有癌症),只有一个非常小的数量丰富的癌症snp与一个特定类型的癌症(表相关联1)。 细节丰富,优势比P值为每个癌症类型,每个社区和每个组织补充表中给出7。
我们探讨了功能影响的癌症snp通过测试社区是否丰富这些snp基因注释也丰富了去生物过程。 在所有组织除了胫骨动脉(ATA),我们发现,增加社区代表的癌症基因snp包含丰富的功能与免疫力,主要属于“主要组织相容性复合体”(MHC)的基因I和II类家庭,和大多数的这些免疫相关基因独联体-eQTLs癌症snp。 基因本体论的一个例子浓缩这个共享社区的全血提出了无花果。1b。其他社区丰富的非特异性的生物过程如RNA代谢过程和DNA结合。 只有两个组织网络提出了一个社区丰富的癌症SNPs和组织生物学途径:皮肤,和粘附在上皮细胞发展成纤维细胞(补充表4).
癌症snp是核心的snp在他们的社区
正如先前所显示的,社区在eQTL网络结构特点,与当地中心或核心snp,中央在他们的社区。 变异单核苷酸多态性通过GWAS找到已经被证明不仅映射到社区相关的生物功能,而且这些社区的核心。22,33作为衡量SNP的中心,我们定义一个“核心得分”等于相对模块化提供的SNP的整体模块化社区(见情商。2在方法部分)。 我们计算核心分数为所有网络中snp和比较核心的分数分布的癌症单核苷酸多态性和单核苷酸多态性与GWAS的性状或疾病无关。 我们发现癌症单核苷酸多态性丰富了更高的核心分数(无花果。2对皮肤和补充图1其他组织)。 跨组织这个结果是一致的,这表明生殖系癌症snp,被他们的社区的中心,许多基因的表达影响参与的生物学过程相关的癌症发展和进展。
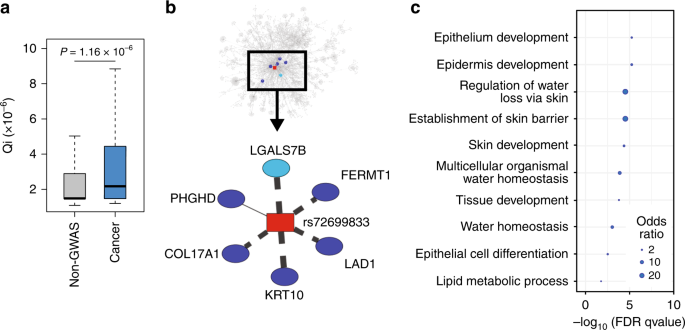
网络GWAS癌症snp的属性。一个核心的分数分布的单核苷酸多态性与癌症发病风险增加GWAS(蓝色)和其他皮肤snp(灰色)。P值是通过使用似然比检验和修剪snp在连锁不平衡。 分布为所有组织网络补充无花果所示。S1。bSNP核心得分高的一个例子:rs72699833,与rs11249433 LD, SNP与患乳腺癌的风险更高。 这个SNP属于社区147年(前面板),这对乳房癌症SNPs和丰富与多种基因参与上皮细胞的发展。 LGALS7B表示但属于另一个社区(107)。 细节的关联提供了补充表8。 虚线表示协会反式完整的线,独联体。 线的厚度对应的关联强度。c基因本体论方面的浓缩为社区147年的皮肤
例如,SNP rs72699833是147年核心SNP在皮肤的社区。 这个SNP与rs11249433 LD与患乳腺癌的风险增加有关(无花果。2b)。通过检查皮肤社区147年,我们发现浓缩与乳腺癌相关的单核苷酸多态性(补充表7基因参与上皮发展)和(补充表4和无花果。2c); 乳腺癌是一种上皮癌细胞,与皮肤并不奇怪。 SNP rs72699833位于染色体1和相关联独联体与PHGDH,一个基因参与新陈代谢的丝氨酸的过表达在某些亚型乳腺癌、宫颈癌、结肠癌和非小细胞肺癌,在这些疾病通常与贫穷相关的结果。37,38,39,40
此外,rs72699833通过eQTL分析相关联反式与其他五个基因:LAD11号染色体上COL17A110号染色体上KRT1017号染色体上LGALS7B19号染色体上,FERMT1在20号染色体(补充表8)。 所有这些基因参与上皮发展,特别是与细胞外基质(ECM)分泌和cell-ECM交互。 大多数这些基因已经被证明是在乳腺癌特异表达或在epithelial-mesenchymal过渡。 的确,LAD1与激进的乳房肿瘤,有关41 COL17A1在乳腺癌和underexpressed在头颈部鳞状细胞癌,肺鳞状细胞癌和肺腺癌42和FERMT1是一种已知的在结肠癌epithelial-mesenchymal过渡的中介。43
癌症snp的优先目标癌症基因
我们预期,癌症snp可能优先相关基因被认为与癌症发展和进步。 我们组建了一个目录的癌基因和肿瘤抑制基因(“癌症基因”)通过使用数据库,包括网络癌症基因版本5.027和宇宙44人口普查(见方法和补充表2).
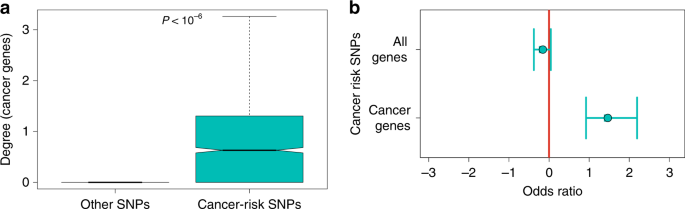
我们测试了癌症snp是否更频繁地与癌症有关的基因比其他基于eQTL snp网络。 我们映射癌症snp giant-connected组件每个十三的组织eQTL网络。 然后我们癌症基因的数量相比癌症SNPs和其他的snp的目标,通过考虑连锁不平衡程度和全球分布(他们相关的基因总数; 见的方法)。 我们表明,癌症snp确实更有可能比预期的目标癌症基因的机会(\ (p < \ \ 1 {0} ^ {6} \)基于1000000重采样)学习时一起网络(无花果。3.一个); 也发现类似的结果在每个组织网络(补充图。S2).
癌症snp是优先位于癌症基因的启动子。一个癌症snp的优先目标癌基因和肿瘤抑制基因在所有组织。 箱形图的分布的肿瘤抑制基因和致癌基因靶向癌症snp和其他单核苷酸多态性。 的P价值是通过使用获得的\ (1 {0}^ {6}\)重采样,通过考虑全球癌症之间分布程度的差异单核苷酸多态性和其他单核苷酸多态性。 这表明癌症基因可能与一个或多个癌症snp,但不是其他eQTL snp。 同样的分析每个组织网络提出了补充图。S2。b癌症snp是优先位于启动子的癌基因和肿瘤抑制基因相对于其他基因。 此图显示了发现癌症的几率比单核苷酸多态性,而不是其他的单核苷酸多态性,在启动子的基因的启动子(上)或癌基因和肿瘤抑制基因的启动子(底部)。 同样的分析对于每个组织eQTL网络提出了补充图。S3.
最后,我们测试了是否癌症snp位于基因的启动子突变在癌症。 在至少1基因表达的13个组织,我们映射的单核苷酸多态性与次要推动者等位基因频率> 5%。 然后我们比较癌症和非癌基因snp映射。 我们发现癌症snp的更不可能比其他snp在非癌基因的启动子区域下降,但癌症snp优先出现在癌基因和肿瘤抑制基因的启动子(无花果。3.b,补充图。S3.).
讨论
它早就知道,生殖系和体细胞突变致癌基因和肿瘤抑制基因驱动开发和发展的癌症。45然而,我们知道癌症遗传因素除了这些著名的“癌症司机”,遗传因素可以影响癌症的自然历史的差异在个人拥有同样的体细胞突变。 全基因组关联研究已经分析了成千上万的个人发现基因变异与患癌症的风险增加相关,但这些落入基因间区域和没有明确的功能与癌症协会的司机。 因此,功能遗传风险之间的联系和癌症发展的机制尚未完全了解。
从GTEx通过使用数据,我们建立了双方的代表生殖系SNP-gene eQTL网络关联,包括顺式和反式-代理eQTLs 13个不同组织中通过使用我们之前使用的扩展方法。33当我们生殖系癌症snp的映射到每一个网络,我们发现癌症snp与癌基因和肿瘤抑制基因的表达水平比预期更大的机会。 这不仅表明在这些癌症突变基因是重要的,但也由监管这些基因变异的基因控制起着重要的作用。 一个自然的假设可能是癌症snp躺在癌基因和肿瘤抑制基因的启动子区域,但是许多GWAS癌症snp的发起人以外,离开这些变异的机制的问题施加自己的影响力。
正如我们之前的报道,SNP-gene eQTL网络组织了高度模块化,监管社区经常丰富的基因进行不同的生物功能。 符合我们之前的分析疾病有关的单核苷酸多态性,22,33我们发现癌症snp的所占比例在个别社区的“核心”,这意味着这些snp在关键职位功能社区癌症单核苷酸多态性可以影响组功能相关基因的表达,从而发挥重大影响关键的生物过程。
尽管GWAS的观察到浓度snp在社区的核心,我们发现疾病相关生殖系snp在eQTL癌症和慢性疾病分布不同的网络社区。 在慢性阻塞性肺疾病(COPD), GWAS snp映射到少量的社区拥有disease-relevant功能。22相比之下,我们发现癌症单核苷酸多态性的分布在大量功能多样化的社区; 这个分布是符合我们明白癌症是一种全身性疾病,影响许多不同的细胞过程。
当我们寻找社区最大的浓缩的癌症snp在所有13 GTEx组织,我们发现这些snp在社区丰富的群体免疫相关基因。 特别是,癌症snp与MHC I和II类基因的表达改变。 MHC基因集中的p-arm 6号染色体,并扮演一个角色在识别pathogen-infected修改和其他类型的细胞(包括肿瘤细胞)和在触发先天和适应性免疫系统。 众所周知,eQTL研究的力量来检测基因型和基因表达的关系取决于小等位基因频率。46,47在这项研究中,我们使用的数据来自13个组织我们可用匹配RNA-seq和基因分型数据在200年或更多的样品; 212年样本大小不同(HRV-heart左心室)和378个样本(SKN-skin)。 即使是最大的样本量不允许我们达到最大功率eQTL检测与low-intermediate等位基因频率(0.1 - -0.2),所以我们的结果很可能是丰富high-intermediate-frequency等位基因(0.2 - -0.5)。 因为MHC地区已知包括许多单核苷酸多态性与高较小的等位基因频率,48我们可能会高估的作用与癌症风险相对于其他相关的基因位点。 进一步的重组率高和高密度snp和MHC基因区域使得关联研究困难。 然而,大多数的eQTL协会在该地区独联体,其中一些已经在先前的研究中发现,有针对性的MHC地区,49,50,51贷款支持我们的研究结果。 调制MHC基因的表达,癌症snp可能修改个人的免疫反应,从而干扰消除变异,癌前细胞。 事实上,那些eQTL-associated免疫反应基因属于MHC I和II类区域已知在大多数癌细胞并影响基因表达下调一些最新的癌症治疗的目标。52,53
除了与免疫反应中观察到所有13个组织协会,癌症snp的所占比例在其他功能上有趣的社区。 例如,单核苷酸多态性与乳腺癌和上皮癌在gwas集群在一个eQTL网络社区在皮肤上网络,丰富的社区和上皮发育有关的基因和细胞外基质分泌。 这些和其他的例子表明,这些单核苷酸多态性的分布和在社区内提供证据的功能意义生殖系单核苷酸多态性与癌症风险和发展相关联。 特别值得注意的是,虽然癌症snp基因表达,组织之间的不同,这些不同的snp通常通过eQTL相关网络社区结构与跨组织的常见功能。 这表明,类似的机制,由组织表达,可能在许多癌症中摄动。 反过来,这很可能会指向常见疾病有关的函数,可以有针对性的治疗。
在13代表eQTLs通过使用由两部分构成的网络组织,我们发现单核苷酸多态性和基因组织了社区反映了基因调控功能相关的基因单核苷酸多态性的影响,证明了GWAS注释、基因本体分析和浓缩的癌症在癌症基因的启动子单核苷酸多态性。 通过相snp映射到这些网络,我们可以开发假设这些snp是如何工作的个人和集体温和的风险和可能使疾病发展。
我们的分析识别重要的监管角色的非编码snp的表演独联体和反式。 Non-genic snp一直会影响基因表达通过改变转录因子结合的网站。 我们也知道non-genic变异可以影响基因表达启动子区域以外的通过修改远程染色质远端之间的相互作用独联体监管元素称为增强剂和它们的目标基因通过修改3 d染色质折叠。54我们的分析表明,监管效果的癌症基因单核苷酸多态性影响癌症基因和其他控制流程与多元化的相关流程,包括发展和免疫反应。 事实上,远程监管影响改变增强剂已被证明在肥胖和帕金森病中发挥作用,55,56和几个例子改变增强剂及其目标基因之间的相互作用,导致肿瘤形成,已被描述。57
这项研究提供了第一个eQTLs利用网络系统分析方法探讨生殖系癌症snp的监管作用。 通过将癌症snp的映射到由两部分构成的网络由两个独联体- - -反式-eQTLs十三组织中,我们表明,癌症snp扮演独特的角色定义这种网络的结构。 癌症snp不仅与癌症有关的基因,但与许多其他与生物功能相关的基因可以导致癌症的发展和进步。 集群的癌症基因单核苷酸多态性和相关成高度模块化的社区提供了一个框架如何将这些snp温和癌症发展的风险。 虽然我们倾向于认为癌症发展的司机,我们的分析表明,这些驱动程序可能会放缓的影响通过与监管这些基因变异的交互,可以增加或减少,罹患癌症的风险。
值得注意的是,这里我们提出的方法还可以用来探索其他snp的功能作用,通过gwas与疾病或其他进程。 虽然eQTL网络的分析不完全基因型和表型之间的桥梁,它提供了一个解释框架,可用于进一步研究疾病的遗传风险和生殖系遗传变异的协同效应。