与心脏代谢特性相关的非保守人类lncRNA的体内功能分析
与心脏代谢特性相关的非保守人类lncRNA的体内功能分析
摘要
与蛋白质编码基因不同,大多数人类长非编码RNA(lncRNA)被认为是非保守的。尽管已经显示lncRNA在小鼠的多种病理生理过程中起作用,但是,人类lncRNA是否具有这种体内功能仍是未知之数。在这里,我们描述了一条整合的管道,以定义非保守人类lncRNA的体内功能。我们首先使用源自与心脏代谢特征相关的人类遗传数据的多种指标来鉴定具有高功能潜力的lncRNA,然后通过整合其在人类中的相关生物途径以及在人源化小鼠模型中共同调控的基因来定义lncRNA的功能和特定的靶基因。最后,我们证明可以在人源化小鼠模型中成功检查人特异性lncRNA的体内功能,
介绍
在过去十年中已经看到了前所未有的进展在我们对人类基因组的认识,解开已成为基因表达的关键调节长非编码RNA(lncRNA)的广泛表达的1,2。LncRNA是长度超过200个核苷酸的转录本,缺乏任何预测的编码潜力。目前,lncRNAs已经在所有的模式生物识别和30,000至60,000人lncRNAs在最近lncRNA注解报道了3,4。与mRNA相比,lncRNA具有更强的组织特异性,并且通常以组织特异性方式发挥作用5。蜂窝实验已经表明lncRNAs参与生物学过程,从细胞增殖,细胞凋亡和营养物的感测到的细胞分化的广谱1,6。lncRNA发挥其功能的主要机制是与转录因子或染色质修饰复合物相互作用,以调节基因转录2。此外,越来越多的lncRNAs已报道在生物体水平研究动物发挥广泛的病理生理过程中的重要作用,7,8,9,10,11,12,表明定义人类lncRNA的功能可以为人类生物学和生理学提供实质性见解。
然而,与蛋白质编码基因,这在很大程度上是哺乳动物保守,绝大多数人lncRNAs的都是非保守的,其中许多人可能是沿袭特定的13,14,15,16。尽管基因组序列的低保护常指一个有限的或缺少的功能,新出现的证据支持,一些非保守人类lncRNAs显然功能,至少在培养的细胞17,18。此外,最近的结构上而不是实例依次保守lncRNAs 19,20提示我们可能还不了解lncRNA保守的模式。目前,评估基因保守性的主要工具是BLAST启发式算法,它依赖于可比序列的连续延伸来进行保守性检测。但是,LncRNAs 除了其序列外,还可能在许多不同的水平上保守21,例如二级或高阶结构,蛋白质结合的短基序,转录但不是转录子起作用的lncRNA的位置22,而BLAST无法有效地识别所有这些。因此,我们目前不知道有多少lncRNA在功能上保守,因此无法使用现有的比较基因组工具来鉴定人lncRNA的特定功能。显然需要一种经过验证的系统来丰富潜在功能的人lncRNA,并推断其在特定生物学过程中的作用,以进行进一步的实验表征。此外,需要合适的实验系统来直接定义非保守人lncRNA的生理功能。细胞培养通常无法对复杂的生理环境进行建模,因此需要一个体内系统来明确定义基因的功能。因此,经常在动物中研究人类蛋白编码基因的生理功能,尤其是小鼠,其中超过90%的人类蛋白编码基因被保守。不幸的是,目前尚没有适合用于研究非保守人lncRNA的体内实验系统。
在这项研究中,我们建立了一条实用的管道,用于研究非保守人类lncRNA的生理功能。我们首先确定一个由肝脏代谢疾病的全基因组关联研究(GWAS)基因座特异性调控的人lncRNA的列表。然后,我们使用来自表观遗传标记和3-D染色质相互作用,肝脏富集,共表达功能预测以及体内代谢环境调节的信息,增强对潜在功能性lncRNA的选择。我们进一步证实,可以在肝脏特异性人源化小鼠模型中定义非保守人类lncRNA的调控和功能。令人鼓舞的是,我们通过鉴定受与肥胖风险增加相关的GWAS基因座调控的非保守人类lncRNA的功能,成功地验证了我们的产品线。
结果
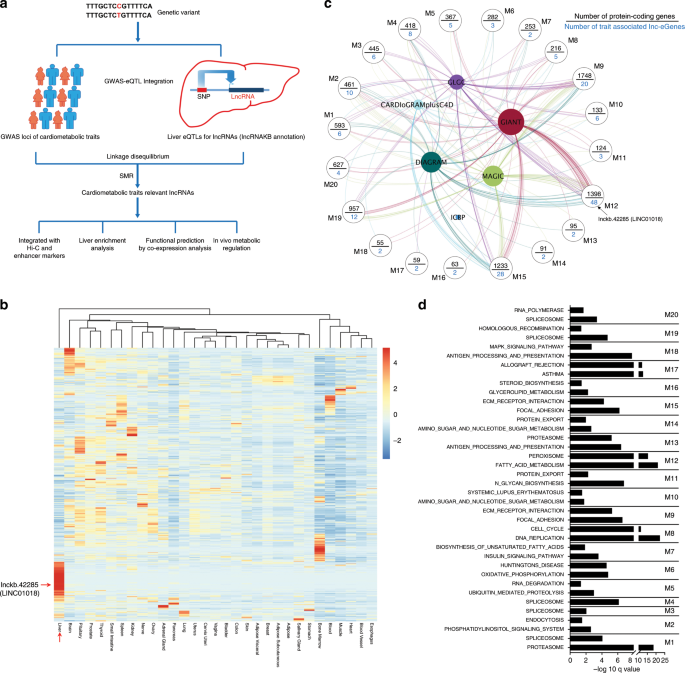
与lnc-eGenes相关的心脏代谢特征的鉴定
GWAS和eQTL信号的整合已导致识别疾病易感性的功能基因23,但这些努力传统上一直集中在蛋白质编码基因上。在这里,我们利用这一关键数据源来识别受GWAS-eQTL共定位基因座调控的功能性lncRNA。
我们对调节肝脏新陈代谢的人lncRNA特别感兴趣,肝脏是新陈代谢的重要器官,并且与一系列心脏代谢疾病高度相关。因此,我们试图系统地绘制由肝脏中的eQTL调控的人lncRNA,这些eQTL与主要的心脏代谢疾病或性状的GWAS基因座共定位(图1a和补充数据 1)。)。尽管已报告的lncRNA数量众多,但只有一小部分被eQTL定位系统地询问,使用基于RNA seq的基因表达谱分析来鉴定eQTL调控的lncRNA基因(lnc-eGene)。为了扩大lncRNA在eQTL计算中的覆盖范围,我们使用最新,最全面的人类lncRNA注释lncRNA知识库(lncRNAKB)(https://doi.org)重新分析了基因型组织表达(GTEx)项目中的人类肝脏RNA序列数据。/10.1101/669994)。然后根据GTEx联合体的基因分型数据计算肝脏表达的lncRNA的cis-eQTL(请参见“方法”)。由于人类基因组中复杂的连锁不平衡,我们应用了SID(基于汇总数据的孟德尔随机化)分析24中实施的HEIDI (依赖仪器的异质性)方法来确定我们鉴定出的lncRNA的eQTL是否在功能上与GWAS信号共定位。总共鉴定了726个独特的lnc-eGenes,它们在肝脏中的eQTL与至少一个名义上显着的GWAS SNP(单核苷酸多态性)共定位(补充数据 1)。),表明它们在肝脏中的表达可能与心脏代谢特征有关。以我们现在的理解相一致,大多数人lncRNAs的都是非保守的13,这些性状相关的LNC-eGenes的平均得分节约大约只有0.12基于30种脊椎动物的多重比对(见“方法”)。
接下来,我们使用了其他一些分析来增强功能性lncRNA的选择(图 1a)。首先,为了获得更多证据,证明与这些性状相关的lnc-eGenes受到其eQTL / GWAS共定位基因座的直接调节,我们整合了H3K4me1和/或H3K27ac ChIP数据以及来自中的高通量染色质构象捕获(Hi-C)数据人类肝脏25(请参阅“方法”)。如补充数据2所示 ,对于44%(726个中的320个)lnc-eGenes,观察到它们的基因区域与eQTL / GWAS共定位基因座之间的空间染色质相互作用,显示了增强子标记。该结果表明这些eQTL / GWAS基因座的相当一部分可能充当调节这些lnc-eGenes表达的增强子。其次,lncRNA在特定组织中的丰富表达可能表明其独特的功能。为了确定已鉴定的lnc-eGenes的肝脏富集,重新分析了GTEx中不同组织的原始RNA-seq数据,并计算了Z值以确定每种lncRNA的表达富集(图 1b和补充数据) 3)。大约有16%的lnc-eGenes的肝Z评分高于2。此外,这些lnc-eGenes的平均肝表达Z评分显着高于随机表达的肝表达基因组(请参见“方法”,表明这些lnc-eGenes表现出更强的肝脏富集表达,并且可能在肝代谢中起特定作用。第三,我们使用肝转录组的共表达网络分析来推断与性状相关的lncRNA的潜在功能。如图1c所示 ,共表达模块识别工具(CEMiTOOL)26用于将这些性状相关的lncRNAs分为不同的功能模块。对每个模块中共表达的蛋白质编码基因的KEGG通路分析表明,这些lncRNA可能在多种代谢途径中起作用,包括脂肪酸代谢,类固醇生物合成,mRNA加工和氨基酸代谢(图 1d和补充数据 4)。
两者合计,我们已经确定了与心脏代谢特征相关的lnc-eGenes的列表,这些基因大多数是不保守的,并且可能在人类肝脏的多种代谢途径中起作用。
研究人类肝脏基因的体内调控的模型
由于上面鉴定的大多数与性状相关的人lncRNA都是非保守的,因此常规小鼠模型不是研究其体内调节和功能的合适工具。为了解决这一挑战,我们试图采用一种新近开发的人源化TK-NOG小鼠模型,在该模型中,肝脏被人类肝细胞重新填充27。人源化小鼠的一个明显的优势是,可以在控制良好的实验条件下,更重要的是,在统一的遗传背景下检查人肝基因表达的变化,这在人类群体中是不切实际的。响应代谢刺激,lncRNA表达水平的此类变化可能提供有关其在代谢中作用的关键线索,从而有助于其下游功能分析。
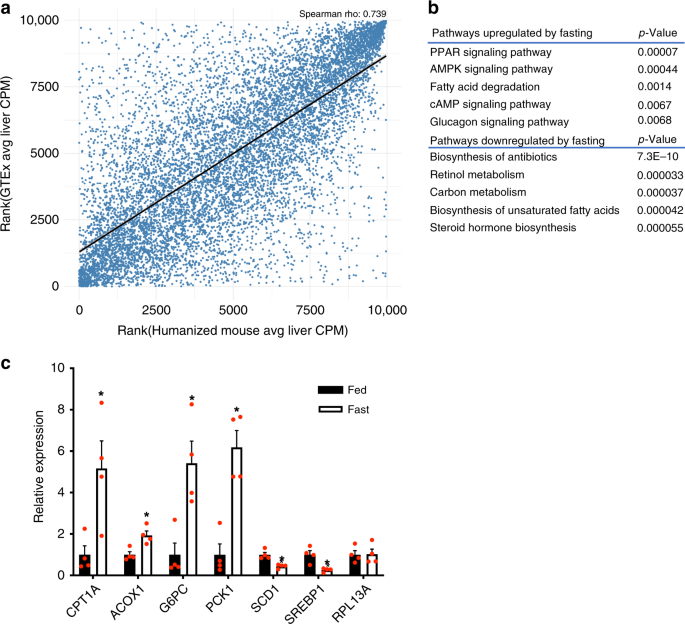
为了评估使用这种人源化小鼠研究非保守的人lncRNA的生理功能的可行性,我们首先检查了人源化肝脏中的人类基因是否维持对代谢禁食等生理反应。我们对24小时停食(禁食)或随意进食(进食)的人源化小鼠肝组织进行了深入的RNA序列分析。为了分析这些嵌合肝脏中的人特异性转录组,首先将RNA-seq读图与人和小鼠的基因组结合,然后仅使用人特异性读物进行进一步分析(请参见方法)。然后,我们对人源化肝脏和人肝中基因表达进行了相关分析,发现人蛋白质编码和非编码基因的表达水平具有可比性(图 2a)。,Spearman的rho为0.739),这表明人源化的肝脏至少可以在转录本水平上忠实地反映人的肝脏生物学。由于对蛋白质编码的基因比对lncRNA基因的理解更了解对空腹反应的肝基因表达,因此我们使用空腹调节的人肝蛋白质编码基因来评估人源化肝脏的功能。途径分析表明,上调的途径包括PPAR信号传导,脂肪酸降解和胰高血糖素信号传导途径,而下调的途径包括碳代谢,不饱和脂肪酸的生物合成和类固醇激素的生物合成途径,这与已知的肝反应完全一致禁食(图 2b)。通过使用人特异性引物的定量实时PCR(qPCR),进一步证实了这些途径中几个关键基因的表达变化(图 2c)。这些结果支持人源化小鼠中的人肝细胞在禁食期间保持其生理功能。而且,通过代谢条件对性状相关的lncRNA的体内调节是推断其生理功能的另一有用信息。值得注意的是,有32个lnc-eGenes在体内通过禁食进行调节(补充数据 5),提供了线索,它们可能在与禁食相关的代谢调节中起作用。
定义与性状相关的lncRNA的体内功能
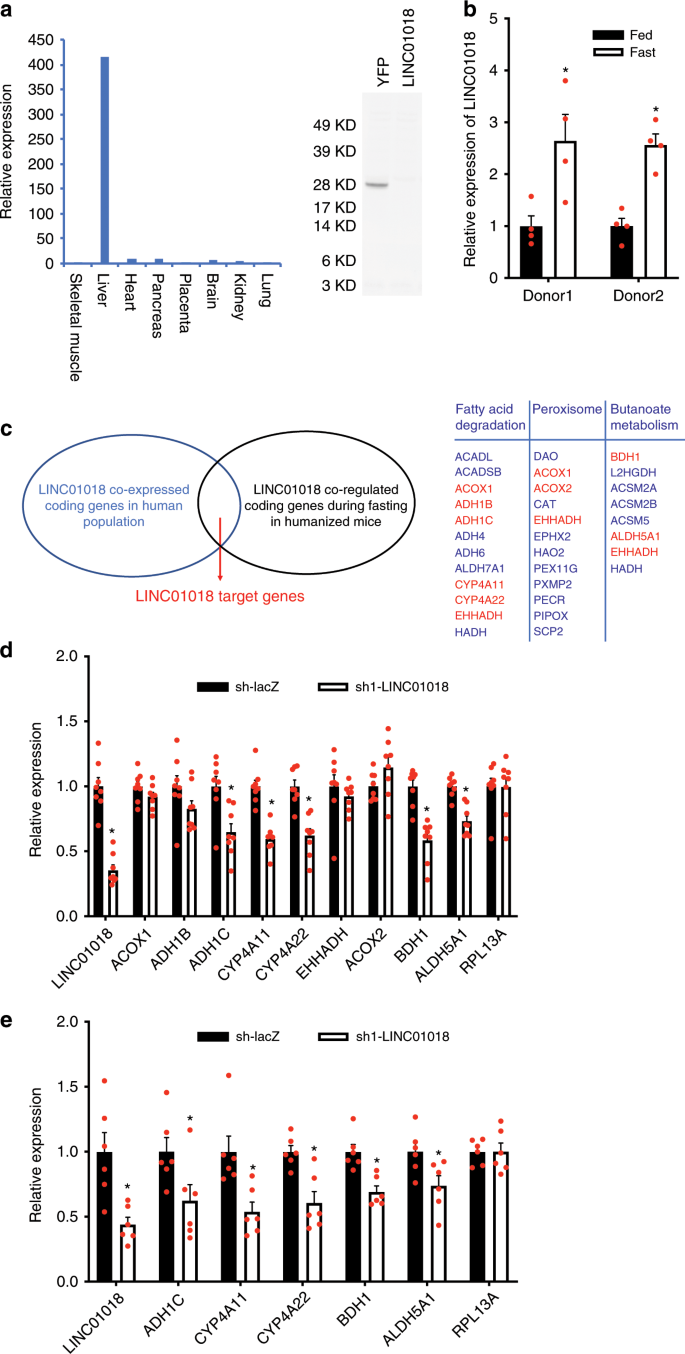
在建立了具有高功能潜力的人lncRNA的选定列表并表征了人肝基因对营养状态表现出适当反应的人源化肝模型后,我们建立了一条实验管线,该管线非常适合询问我们已鉴定出的性状相关的功能意义lncRNA。在已鉴定的lncRNA中,我们对lnckb.42285(LINC01018)特别感兴趣,lnckb.42285是染色体5上的非保守基因间lncRNA(补充图 1a)。来自我们管线的多条证据表明LINC01018可能在代谢中起作用:GWAS-eQTL整合分析显示,肝脏中LINC01018的表达水平与BMI和胰岛素敏感性指数有关(补充数据 1)。); 在人类肝脏中进行了表观遗传标记和3-D染色质相互作用分析,发现LINC01018的对应GWAS-eQTL共定位基因座同时包含H3K27ac和H3K4me1活性增强子标记,并与LINC01018的基因组区域相互作用(补充数据 2);GTEx数据集中的人体组织表达谱(图 1b)以及使用人体组织cDNA面板的qPCR结果(图 3a)显示LINC01018的表达在肝脏组织中高度富集。尽管在LINC01018上以计算方式检测到开放阅读框(补充图 1b),但是使用多种算法(补充图1c)和体外翻译分析(图 3a)进行编码潜力分析 )证明LINC01018是非编码的。有趣的是,基于我们的RNA-seq分析(补充数据5)和随后的qPCR分析(图 3b),通过在人源化肝脏中禁食,LINC01018被强烈上调 。由于人类基因表达的调节有时会受到遗传背景的强烈影响,因此我们还检查了由第二位独立供体的人肝细胞产生的人源化小鼠中LINC01018的表达,发现禁食对LINC01018的上调得到了很好的维持(图。 3b)。人肝细胞中LINC01018的估计平均拷贝数约为74(补充图 1d),人源化肝组织的亚细胞分级分离显示LINC01018分布在细胞质和细胞核中,并且在细胞质中分布更多(补充图 1e)。总的来说,LINC01018是一种与代谢性状相关,非保守且富含肝脏的人lncRNA,可能在与禁食相关的肝脏代谢过程中发挥作用。
a左:使用来自Clontech的人cDNA组织板通过实时定量PCR分析了LINC01018在骨骼肌(设置为1),肝脏,心脏,胰腺,胎盘,脑,肾和肺等人体组织中的相对表达水平。右:使用YFP和LINC01018的RNA进行体外翻译分析。b LINC01018在人源化小鼠( 每组n = 4)中禁食时的肝表达,该禁食是由来自两个不同供体的人原代肝细胞生成的。C左:图示说明了识别潜在LINC01018靶基因的策略。右图:在由LINC01018相关蛋白编码基因过度代表的主要途径中,潜在的LINC01018目标基因以红色突出显示,并且在人源化小鼠中与LINC01018一起禁食也对其进行调节。d对照和LINC01018敲除(KD)人源化小鼠肝脏中LINC01018及其潜在靶基因的表达水平分别接受了针对LacZ(LacZ sh,n = 8)和LINC01018(LINC01018 sh1,n = 8)的shRNA腺病毒。e对照(LacZ sh,n = 6)和LINC01018 KD(LINC01018 sh1,n = 6)停食24小时后,使用来自第二个供体的人原代肝细胞进行人源化小鼠。(b,d,e)中的数据代表平均值±SEM,* p <0.05,两尾未配对的学生t检验。(a,b,d,e)的源数据在源数据文件中提供。
前述共表达模块分析已将LINC01018置于涉及人类肝脏中各种关键代谢途径的模块中(图 1c,d和补充数据 4)。为了查明与LINC01018具体相关的基因和途径,我们接下来进行了聚焦的lncRNA-mRNA相关分析。使用来自GTEx的人肝RNA序列数据,计算LINC01018的表达与所有肝表达的mRNA之间的成对Pearson相关性。然后,使用前300个相关编码基因的列表来定义丰富的KEGG途径。如图3c所示 ,该分析发现LINC01018可能在脂肪酸降解,过氧化物酶体和丁酸酯代谢中起作用,证实了图1c,d中 LINC01018共表达模块M12所代表的最重要的途径 (补充数据 4)。这些途径在禁食期间在肝脏中被激活,这是一种已知会增加LINC01018表达的疾病。
由于大多数报道官能lncRNAs,包括那些先前我们已经表征10,11,28通过调节基因表达执行它们的功能,它是可能的LINC01018也可能调节它的相关的基因的表达。同时,LINC01018被禁食强烈上调,表明它可能有助于这些禁食诱导基因的表达增加。因此,我们在人肝中重叠了与人肝中与LINC01018具体相关的基因,并在人源化肝中鉴定出禁食上调的人基因,并将其定义为潜在的LINC01018目标基因(图 3c)。)。为了测试这些基因是否可以在体内被LINC01018调节,我们使用腺病毒在人源化小鼠中表达靶向LINC01018的短发夹RNA(shRNA),该策略成功地将LINC01018的表达降低了70%(图 3d)。因此,我们发现虽然人ACOX1,ADH1B,EHHADH和ACOX2的表达水平不受影响,但敲除LINC01018导致脂肪酸代谢的ω-羟基化途径中ADH1C,CYP4A11和CYP4A22的表达降低。在丁酸代谢途径中与BDH1和ALDH5A1相同(图 3d)。我们注意到,LINC01018的最邻近基因NSUN2和SRD5A1的表达不受LINC01018敲低的影响(补充图 1f)。),表明LINC01018不太可能顺式作用。在人源化小鼠中使用第二个shRNA敲低LINC01018表现出对这些基因表达的相似作用,支持我们观察到的调节作用由LINC01018特异介导(补充图 1g)。为了检查LINC01018对基因表达的调控是否受到遗传背景的影响,我们在第二只独立供体的肝细胞产生的人源化小鼠中敲低了LINC01018,发现其中的ADH1C,CYP4A11,CYP4A22,BDH1和ALDH5A1的表达水平小鼠同样减少(图 3e)。尽管lncRNA可能在具有不同遗传背景的个体中发挥独特作用,但我们的数据表明,由我们的策略定义的LINC01018对基因表达的具体调控可能与遗传背景的影响无关。
综上所述,这些数据表明LINC01018调节脂肪酸代谢中基因的表达。通过在与lncRNA有关的实验条件下(例如本例中的禁食)也对lncRNA相关的top途径中的基因与受调控的基因相交,来定义lncRNA的潜在靶基因是一种有用的策略。更重要的是,我们的分析在此根据人类数据定义了人类lncRNA的推定靶基因,这将有助于鉴定具有直接人类影响的靶标,还有助于避免人源化肝脏引入的潜在伪像。
LINC01018的功能无法在体外概括
培养的原代人肝细胞(PHH)被广泛用于研究人肝基因的调控,我们感兴趣的是测试LINC01018的功能能否在此体外系统中得到概括。实际上,在培养的PHH中LINC01018的相对表达保持在与人源化肝脏相当的水平。由于肝脂肪酸降解途径中的LINC01018相关基因通常在禁食期间以PPARα依赖性方式被激活29,因此我们认为用PPARα激动剂治疗PHH可能能够诱导脂肪酸降解途径和LINC01018中基因的表达。与以前的报告一致30用PPARα激动剂GW7647治疗PHH强烈上调了CYP4A11和CYP4A22的表达(补充图 2a)。但是,在此条件下未观察到LINC01018表达的诱导。此外,普通应力对PHH中LINC01018的表达没有影响(补充图 2b)。然而,我们试图测试LINC01018敲低在培养的PHH中的作用。虽然图3d中使用的相同shRNA 成功地将LINC01018的表达阻断了70%,但其靶基因的表达在PHH中不受影响(补充图 2c))。这些结果表明,培养的PHH不适合研究我们在人源化小鼠中观察到的LINC01018的调控和功能,突出了体内模型对研究人lncRNA在代谢调控中的功能的重要性。
HuR在体内介导LINC01018的调节作用
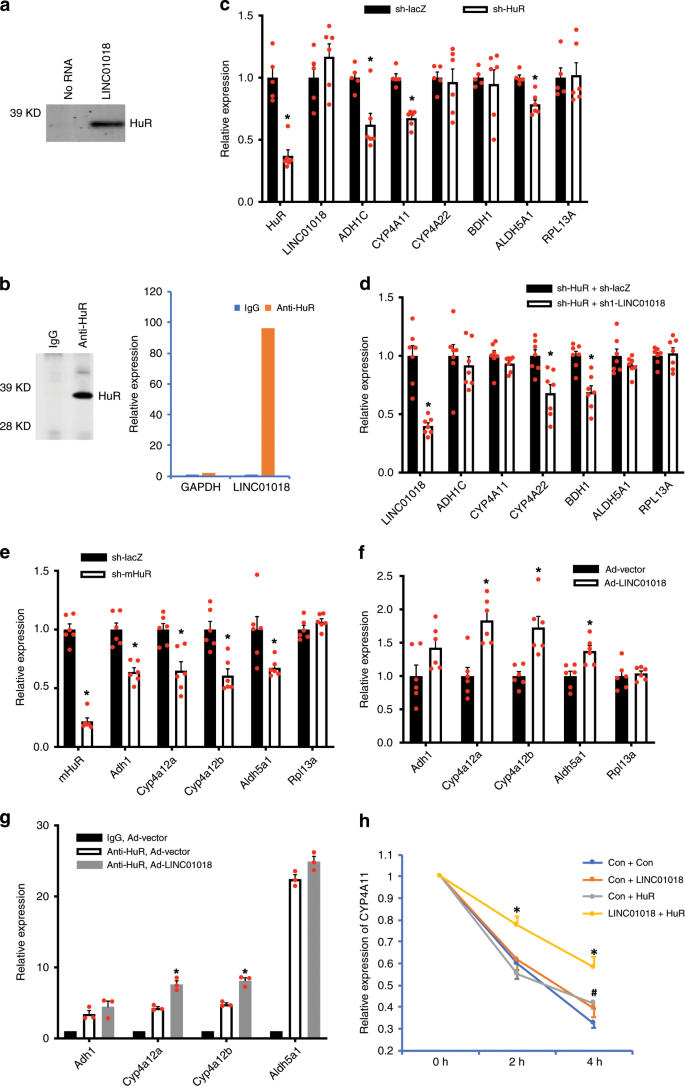
为了探索LINC01018调节体内基因表达的分子机制,我们进行了lncRNA下拉分析,以鉴定富含LINC01018的人类蛋白质。该策略成功地鉴定了几种参与RNA代谢的RNA结合蛋白(补充数据 6)。其中,我们对HuR特别感兴趣,HuR是一种经过充分研究的RNA结合蛋白,可调节mRNA的稳定性和RNA代谢的其他步骤31。为了验证HuR和LINC01018之间的特异性相互作用,我们通过使用抗HuR抗体的免疫印迹证实了质谱分析结果(图 4a)。)。我们进一步使用HuR抗体进行RNA免疫沉淀,以确定人源化小鼠肝组织中内源性HuR与LINC01018之间的相互作用。如图4b所示 ,LINC01018高度富集了HuR免疫沉淀,而没有富集丰富的对照转录物甘油醛3-磷酸脱氢酶(GAPDH)。这些数据支持LINC01018在体内与HuR特异性相互作用。
使用抗HuR抗体对结合生物素化LINC01018或对照的人源化肝组织提取物中蛋白质的免疫印迹分析。b左图使用抗HuR抗体对人源化肝组织免疫沉淀物中的蛋白质进行抗HuR免疫印迹分析。正确,使用抗HuR抗体的人源化肝组织免疫沉淀物中的甘油醛3-磷酸脱氢酶(GAPDH)和LINC01018 RNA水平。c。禁食 24小时后,人肝癌对照(LacZ sh,n = 5)和HuR KD(HuR sh,n = 6)肝脏中的人类HuR和LINC01018靶基因的表达水平。d在接受对照(LacZ shRNA)和HuR KD或LINC01018 KD和HuR KD腺病毒的人源化小鼠肝脏中的基因表达( 每组n = 7)。e在接受对照(LacZ shRNA)或小鼠HuR KD腺病毒的野生型小鼠肝脏中的基因表达( 每组n = 6)。f在接受对照或LINC01018过表达(OE)腺病毒的野生型小鼠肝脏中的基因表达( 每组n = 6)。g使用接受对照或LINC01018 OE腺病毒的野生型小鼠的肝组织裂解液,抗HuR免疫沉淀物中的相对RNA水平( 每组n = 3)。(c – g中的数据)代表平均值±SEM,* p <0.05,两尾未配对的学生t检验。h在放线菌素D处理0、2和4小时后,人CYP4A11在293 A细胞中的相对表达。数据代表三个独立实验的平均值±SEM,* p <0.05为“LINC01018 +的HuR”与“精读+ CON”相比,#p <0.05为“精读+的HuR”与“精读+ CON”相比,双尾未配对的学生的t检验。(a – h)的源数据在源数据文件中提供。
由于HuR是基因表达的众所周知的转录后调节剂,因此我们认为,如果LINC01018的调节功能是由HuR介导的,则LINC01018目标基因的转录不应受到LINC01018敲低的影响。为了测试这一点,我们使用抗RNA聚合酶II抗体进行了染色质免疫沉淀(ChIP)分析,以确定人源化肝脏中人ADH1C,CYP4A11,CYP4A22,BDH1和ALDH5A1的转录活性。尽管RNA聚合酶II在这些基因的转录起始位点高度富集,但敲除LINC01018在人源化小鼠中未观察到它们富集的变化(补充图 3a)。),表明LINC01018通过转录后机制发挥调节作用,HuR可能参与了这一过程。为了直接测试这种可能性,我们使用腺病毒在人源化小鼠中递送特别靶向人HuR的shRNA,然后检查了LINC01018靶基因的表达。当人源化小鼠肝脏中超过60%的人类HuR表达被抑制时,五个LINC01018调控基因ADH1C,CYP4A11和ALDH5A1中的三个表达水平显着降低(图 4c),这表明HuR可能介导LINC01018的某些调节作用。为了进一步测试LINC01018对HuR的功能依赖性,我们同时敲除了人源化小鼠肝组织中的LINC01018和HuR。如图4d所示 当阻断人HuR的肝表达时,敲低LINC01018对ADH1C,CYP4A11和ALDH5A1的表达没有影响,而对CYP4A22和BDH1的表达的影响仍然很大。这些数据支持LINC01018至少部分以依赖于HuR的方式运行。
尽管无法基于序列相似性鉴定出LINC01018的小鼠同源物(补充图 1a),但lncRNA已被提议以其他水平保守,包括结构,功能或基因组位点21。为了探索可能从小鼠基因组的同义区域转录LINC01018小鼠同源物的可能性,我们在Nsun2和Ube2ql1之间的该区域中搜索了所有潜在的未表征的转录本(补充图 3b),并确定了它们在小鼠肝脏中的表达水平。在十个假定的带注释的lncRNA中,只有两个(Gm48819和Gm18313)在小鼠肝脏中可检测到。然而,它们均未显示出明显丰富的HuR免疫沉淀(补充图 3c)。),也没有像LINC01018一样对小鼠肝脏中的禁食做出反应(补充图 3d)。鉴于它们在小鼠肝脏中的表达水平非常低(两者的估计拷贝数/细胞均小于0.5,而人类肝细胞中LINC01018的拷贝数却不到74),Gm48819和Gm18313不太可能成为LINC01018的小鼠同源物。
由于无法立即鉴定LINC01018的小鼠同源物或根本不存在它们,我们试图测试观察到的LINC01018结合蛋白HuR的功能在人和小鼠之间是否保守,以及LINC01018的异位表达是否调节其活性。普通小鼠中的小鼠HuR。为此,将携带靶向小鼠HuR的shRNA的腺病毒递送至普通小鼠,并测定人ADH1C,ALDH5A1和CYP4A11的小鼠同源物的表达水平。当抑制小鼠肝脏中超过70%的HuR表达时,小鼠Adh1,Aldh5a1,Cyp4a12a和Cyp4a12b的表达水平显着降低(图 4e),表明HuR对LINC01018靶基因的调控在小鼠中是保守的。接下来,将表达LINC01018全长cDNA的腺病毒送入普通小鼠体内,使其在小鼠肝脏中表达LINC01018的水平与人肝组织中的水平相当,HuR免疫沉淀法明确表明LINC01018可以有效结合小鼠HuR(补充图 3e)。更重要的是,我们发现表达LINC01018的小鼠肝脏组织中的小鼠Cyp4a12a,Cyp4a12b和Aldh5a1的表达与接受对照病毒的小鼠相比有实质性的诱导(图 4f)。),表明LINC01018可以调节小鼠肝脏中的HuR活性。为了直接测试LINC01018是否调节小鼠的HuR活性,我们进行了HuR免疫沉淀测定,发现LINC01018的表达显着增强了小鼠肝脏中HuR与Cyp4a12亚型的mRNA之间的相互作用(图 4g)。为了清楚地证明LINC01018-HuR复合物对mRNA稳定性的协调调节,我们在HEK293A细胞中过表达人CYP4A11和LINC01018和HuR的组合,并进行了放线菌素D处理以确定RNA降解的速率。如图4h所示 ,而单独的LINC01018或HuR的过表达对CYP4A11 mRNA的稳定性影响极小,而同时表达两者均显着减慢了CYP4A11的降解。这些数据进一步支持LINC01018通过调节HuR的活性来调节其靶基因的mRNA稳定性。
最后,为进一步确定LINC01018在人类中的病理生理相关性,我们分析了与非酒精性脂肪肝疾病(NAFLD)相关的人类肝脏RNA-seq数据,发现LINC01018在NAFLD患者肝脏中的表达水平显着降低与健康对照对象相比(补充图 4a)。更有趣的是,对NAFLD患者进行低碳水化合物饮食干预会显着增加肝脏中LINC01018的表达,并伴有LINC01018目标基因的上调(补充图 4b)。)。这些数据表明,LINC01018可能有助于脂肪性肝病患者的保护性代谢。实际上,当用高蔗糖饮食挑战肝脏中强烈的新生脂肪生成时,与对照小鼠相比,表达LINC01018的常规小鼠表现出较低的甘油三酸酯和游离脂肪酸循环水平(补充图 4c)。
两者合计,这些结果表明,LINC01018,一种非保守的人lncRNA,调节HuR的保守功能,以调节代谢基因的表达。尽管不能完全排除小鼠肝脏中存在与LINC01018类似功能的lncRNA的情况,但我们的数据支持非保守的人lncRNA可能在能量代谢中进行人特异性调控。
讨论区
目前,大多数人lncRNA被认为是非保守的,这使得鉴定潜在的功能性RNA并进一步定义其体内功能极为困难。在这里,我们开发了一个管道来同时解决这两个挑战。使用该管道,我们提供了原理上的证明,即此类lncRNA LINC01018在体内调节肝脏代谢基因的表达。我们进一步证明了LINC01018与HuR相互作用以发挥其功能。有趣的是,尽管我们观察到的HuR对代谢基因的调控作用在小鼠和人类中均得以保留,但LINC01018仅在人类中表达,构成了另一层微调机制。因此,通过建立完全集成的端到端实验管道,
目前,人类lncRNA的有限的序列保守性在定义其在人类生理学中的功能方面提出了重大挑战。但是,由于我们对lncRNA功能的了解仍然是基本的,因此我们可能尚不了解lncRNA的保守性,而lncRNA功能的保守性可能无法通过当前主要为蛋白质编码基因设计的工具进行有效评估。因此,具有功能的lncRNA的百分比可能远远大于当前基于序列的保守性分析所建议的百分比。但是,由于我们目前缺乏使用序列信息来识别功能性lncRNA的有效策略,我们付出了巨大的努力来鉴定可能起作用的人lncRNA,并努力提供尽可能多的功能推断,以简化其下游分析。我们首先系统地筛选受与代谢性疾病相关的eQTL-GWAS基因座调控的肝脏富集的lncRNA。肝功能与这些代谢紊乱的病理生理高度相关,并且已证明许多肝脏富集的lncRNA可调节小鼠肝和全身代谢的各个方面10,11,12,28。同时,我们进行了其他分析,以增强对这些与性状相关的lncRNA功能的信心。例如,从我们的分析中得出的多条证据表明,LINC01018可能在代谢中发挥作用,包括其通过GWAS-eQTL对的调控以及肝脏表观遗传标记和3-D染色质相互作用的额外支持,在肝脏中的表达富集,并将其与参与脂肪酸代谢途径及其禁食调节的基因相关联。应当注意的是,在纯人类遗传背景下以及在良好控制的实验条件下,在人源化小鼠体内通过体内代谢环境对这些lncRNA的调控信息,对于lncRNA的下游体内功能分析应具有很大的指导意义。
研究人类lncRNA的最实际挑战可能是缺乏定义其生理功能的实验系统。由于大多数人类lncRNA是人类或灵长类动物特异性的,因此通常只能在人类系统中进行研究。然而,大多数源自人的肝细胞系或原代肝细胞通常表现出与人体中天然生理状态下存在的肝细胞明显的偏离。为了应对这一挑战,我们采用了肝脏特异性的人源化小鼠模型来研究人类lncRNA,并成功鉴定出非保守人类lncRNA的特异性功能。有趣的是,我们无法在原代肝细胞中检测到该lncRNA的类似功能影响,这是我们和其他人在表征小鼠lncRNA的过程中反复观察到的现象11,12,28。这些发现共同支持体内系统对于定义非保守人类lncRNA基因的生物学作用至关重要。
当然,人源化肝脏不是人的肝脏,显然有其局限性。与其他器官类似,肝脏由多种细胞类型的结构化组织组成,但是在当前模型中,只有肝细胞被部分“人源化”。在这种嵌合肝中,小鼠肝细胞或其他细胞的比例可能会影响结果。另外,人源化的小鼠不适合研究其功能需要人激素系统的人lncRNA。该模型还具有研究肝脏早期成熟的局限性,因为它仅在小鼠成年后才衍生。众所周知,免疫力和新陈代谢高度相关32但是用于人类肝细胞移植的小鼠免疫缺陷,不适合研究在这方面起作用的lncRNA。目前,我们的人源化肝脏模型仅是人类肝脏的近似模型,可能仅适合于研究人类肝脏代谢的某些方面。但是,这种近似仍然值得努力。人源化小鼠中的人类肝细胞表达数千种人类特异性lncRNA,其中许多只能在肝脏的生理环境中维持。此外,我们注意可能由这种近似引起的实验伪像,并已采取措施来减轻这种可能性。例如,我们已经从人类数据中提取了所有lncRNA靶标,并将潜在的lncRNA调控基因抑制为人类数据所支持的那些基因,例如基于GTEx数据资源的基因共表达分析。与任何其他模型系统相似,肝脏特异性人源化小鼠已经并将继续得到改进,以更可靠地模拟人肝。可以同时移植肝细胞和造血细胞,以研究需要肝细胞与血淋巴样细胞之间相互作用的过程33。理论上,其他代谢器官(例如脂肪和肌肉)也可以在同一只小鼠中同时被人源化,这将建立一个更完整的系统来研究lncRNA在器官沟通中的作用。
非保守人类lncRNA的数量已经超过了mRNA的数量,对这些lncRNA的功能分析可以提供许多有关人类生理学和疾病的基本见解。例如,两个非保守人类lncRNAs最近显示出在细胞分别调节脂肪细胞和胆固醇代谢17,18。目前的研究进一步证明了人类特异性lncRNA在体内的生理相关性。GWAS研究通常将基因组位点分配给蛋白质编码基因,并且常常忽略了lncRNA的作用。我们已经在这里表明,性状相关的人类lncRNA调节可能在其相关疾病中起作用的代谢途径。其他与性状相关的lncRNA的进一步功能研究可以提供对常见人类疾病遗传基础的重要见解。最终,对非保守人类lncRNAs如何影响基因表达和细胞生物学的更深入的遗传和分子理解可以转化为人类疾病更好的病理生理特征以及更有效和个性化的疗法。
方法
RNA-seq分析流程
正常人群的肝脏RNA-seq数据可从GTEx项目下载。在序列读取档案(SRA)中分别从SRP186450和SRP126075检索了14例健康对照者和15例非酒精性脂肪肝疾病(NAFLD)患者以及7例低碳水化合物饮食干预前后的NAFLD患者的肝RNA-seq数据集。经过质量控制(FastQC:https ://www.bioinformatics.babraham.ac.uk/projects/fastqc/ )和修整(TrimGalore:https ://www.bioinformatics.babraham.ac.uk/projects/trim_galore/ )之后,将fastq文件与使用GRCh38基因组创建的索引和使用HISAT2 34的lncRNAKB注释参考对齐。桑班巴sort 35用于压缩和排序生成的sam文件的文件,使用Subread程序包37中的 featureCounts 36生成每个样本的表达计数水平。去除低读数对齐(<100万)的异常样本。 半数样本中CPM 38 <2的蛋白质编码基因和CPM <1的非编码基因被排除在进一步分析之外。
lncRNA的肝脏eQTL发现
使用GTEx v7 RNA序列数据和WGS基因分型数据(n = 118个样本)39进行肝脏eQTL发现。GTEx RNA-seq数据使用上述自定义注释进行了定量。基因型样本主要来自具有欧洲血统的个体,但也包括具有非洲或亚洲血统的个体。基因分型数据从GRCh37提升到GRCh38。我们的管道紧随GTEx联盟(http://github.com/broadinstitute/gtex-pipeline)使用的eQTL发现管道。将表达数据TMM归一化40,并根据上述临界值过滤基因。使用FastQTL 41进行Cis-QTL分析测试该基因周围1 Mb以内的所有SNP。分析中包括前三个基因分型主要成分(PC),15个表达残基的概率估计(PEER)42个因素,性别和基因分型平台作为协变量。丢弃多等位基因SNP,并使用dbSNP v151数据库43注释其余SNP 。
为了解决lncRNA 顺式 -eQTL 的可靠性,使用了一种排列策略。我们对FastQTL中的数据执行了自适应排列遍历(介于1000和10,000之间的排列)。使用与初始cis -eQTL分析相同的协变量和参数来运行排列。选择所有经验排列p值≤0.05(n = 1701)的lncRNA基因进行进一步分析。另外,仅将顺式 -eQTL p值≤5×10 -4(n = 146,062)的SNP-lnc-eGene对进一步选择用于SMR分析。
GWAS和eQTL信号的共定位分析
使用SMR程序中实现的SMR(基于汇总数据的孟德尔随机化)和HEIDI(相关工具中的异质性)方法来测试基因表达与性状之间的关联以及GWAS和eQTL信号的共定位。特别是,HEIDI在顺式eQTL区域中使用多个SNP来区分多效性与连锁性,pHEIDI> 0.05表示非异质性,因此是共定位的。简要地,下载了六个代谢组进行GWAS荟萃分析的主要代谢性状的GWAS汇总数据(补充数据 1),并格式化为CNS基因组学网站(http://cnsgenomics.com)上指定的.ma格式。/ software / smr /)。eQTL摘要数据由146,062个顺式 -eQTL组成(p选择的1694个lncRNA中的r-value≤5 ×10 -4,经验排列p值≤0.05。FastQTL的结果被格式化为BESD格式。然后分别使用GWAS荟萃分析摘要数据对29个心脏代谢特征中的每一个进行单独分析(补充数据 1),默认的cis窗口为2000kb,将peQTL 的顶部SNP 设置为5×10 -4,并使用1000个基因组数据作为参考。44。考虑到由于来自GTEx的肝脏样本数量较少,我们的eQTL计算的统计能力有限,因此我们最初将Snc分析中的lncRNA保留在标称pSMR <0.05而pHIEDI> 0.05的情况下,并使用下文所述的其他下游分析来进一步选择功能性lncRNA。
表观遗传标记和Hi-C染色质相互作用分析
使用Hi-C Pro软件46分析来自单个样品(SRX641267)45的5次人类肝脏Hi-C测序数据的读数使用默认条件。检索有效的具有空间相互作用的基因组片段对。从人类肝脏的ChIP-seq分析中提取的H3K27ac(ENCSR678LND和ENCSR230IMS)或H3K4me1(ENCSR489DUV,ENCSR218ZMU,ENCSR642HII和ENCSR111OHT)组蛋白标记富集是从ENCODE下载的。为了找到eQTL SNP,H3K27ac / H3K4me1和lncRNA之间相互作用的证据,我们要求Hi-C对的一个片段包含在我们的目标库中鉴定出的eQTL SNP,并且与一个或多个H3K27ac / H3K4me1富集峰重叠。 Hi-C对的另一个片段与726个lncRNA基因之一重叠至少一个碱基对。发现320个基因具有至少一对这样的Hi-C对的支持。
养护分析
可以在lncRNAKB的网站上找到详细的方法(http://psychiatry.som.jhmi.edu/lncrnakb/methods.php)。简而言之,使用bigWigAverageOverBed和cons30way(hg38)轨道(均从UCSC基因组浏览器下载)分析了lncRNA的外显子保守性。此曲目显示了30种脊椎动物物种的进化保护的多重比对和测量。使用针对lncRNA的lncRNAKB GTF注释文件创建了外显子级BED文件。我们在转录本中合并了重叠的外显子,以避免重复计数重叠碱基对的保守得分。对于每个外显子,bigWigAverageOverBed函数将计算所有碱基对的平均保守评分。然后从每个lncRNA基因的所有外显子计算出平均得分。与726个性状相关的lnc-eGenes的平均平均保守评分为0.12。
肝富集
富肝基因定义为肝脏表达z-分数> 2的基因。使用我们的RNA序列分析管线重新分析GTEx中所有可用的样品。对于每个组织,计算所有样品的平均CPM,并计算每个基因的组织特异性z评分。
为了测试我们的性状相关的lnc-eGenes是否总体上具有更强的肝脏富集表达,计算了所有性状相关的lnc-eGenes的平均肝z得分。然后用随机抽样的肝表达基因列表重复10,000次此过程,并替换为与我们与性状相关的lnc-eGenes列表相同大小的列表。对于每次重采样,均会收集平均Z分数以创建经验肝脏Z分数分布。将与性状相关的lnc-eGenes的平均z得分与该分布进行比较以获得肝脏富集的经验p值。
使用网络方法预测lncRNA的功能
为了鉴定使用SMR分析鉴定的726个lncRNA的潜在功能,使用CEMitools R软件包26自动执行基因共表达网络分析,并使用从MSigDB 47下载的KEGG途径进行途径富集。根据两个标准进一步过滤鉴定出的簇:(1)从肝eQTL-GWAS分析(lnc-eGenes)中鉴定出至少一种lncRNA。(2)簇中的基因至少富集了一条KEGG途径,q值<0.05。这导致鉴定出29个模块/基因簇。
聚焦的lncRNA-mRNA相关性分析
为了找到感兴趣的lnc-eGene的潜在特定靶基因,将来自GTEx的人肝RNA-seq数据用于聚焦的lncRNA-mRNA相关性分析。简而言之,对于所有肝脏表达的编码基因(一半样本中的cpm≥2),在lnc-eGene表达与mRNA表达之间计算成对的皮尔逊相关性。计算了前300个相关编码基因的KEGG途径富集,并将在前3个途径中富集的基因视为lnc-eGene目标基因的潜在候选者。
人源化小鼠肝脏的RNA-seq分析
将来自lncRNAKB的人类注释与Refseq小鼠注释结合在一起,以构成人和小鼠的杂交基因组注释,以分析来自人源化小鼠的嵌合肝脏的RNA-seq数据。每个注释的重叠群首先以“ human_”和“ mouse_”为前缀,具体取决于源生物。我们还遵循相同的步骤,并生成了组合的FASTA文件以进行索引。使用我们的RNA-seq流程处理了八个人源化的小鼠RNA-seq样品(四个来自禁食小鼠;四个来自喂食小鼠)。一旦通过featureCounts生成了表达表,就分离了人类基因,以进行进一步的下游分析。DESeq2 R包装48然后用于计算进食和禁食小鼠之间差异表达的基因。
动物实验
所有动物实验均根据NHLBI动物护理和使用委员会或中央实验动物研究所(CIEA)的动物护理委员会的批准进行。基于数据收集过程中或其他健康问题(例如抗伤口或感染)的可见肝结构异常的既定标准,从实验中排除了动物数据。根据代谢参数的可变性,在以前的研究的基础上,使用实验室和中试实验中的类似测定方法确定组大小。实验者对治疗组不知情。
从Taconic Biosciences获得了TK-NOG小鼠,其中在高度免疫缺陷的NOG小鼠的肝脏中表达了小鼠白蛋白启动子下的单纯疱疹病毒1型胸苷激酶(TK)转基因。TK将抗病毒药物更昔洛韦(GCV)转换为有毒产品,可在体内选择性消除TK阳性细胞。冷冻保存的原代人肝细胞获自Thermo Fisher Scientific(第一供体)或BioIVT(第二供体)。如先前所述制备人源化的TK-NOG小鼠27。简而言之,在8–9周龄的TK-NOG小鼠腹腔注射GCV,剂量为25 mg / kg。一周后,50–μl的体积为1×10 6悬浮于HBSS溶液中的人类原代肝细胞通过脾内注射进行移植。移植后8-12周,测量小鼠血清中人白蛋白,作为人肝细胞置换程度的指标。血清人白蛋白水平高于0.5 mg / ml的人源化TK-NOG小鼠用于实验,其中q-PCR可以可靠地检测人肝基因。为了进行禁食研究,产生了人源化的小鼠,并在CIEA进行了实验。其余研究的人源化小鼠在NHLBI进行生产和分析。对于禁食研究,在组织收获之前,允许人源化的TK-NOG小鼠自由进食或戒断24小时的食物。
在8周大时从杰克逊实验室购买了雄性C57BL / 6(B6)小鼠,每笼饲养3-5只小鼠,可以自由饮水和正常食物(NIH-31),使动物适应环境适应环境实验前10-14天。将共同饲养的小鼠组按照年龄和体重随机分配到实验组中。对于高蔗糖饮食喂养实验,在腺病毒注射后3天,给小鼠喂养高蔗糖饮食(Research Diets Inc,目录:D08020801)4天,然后收集血浆和肝组织用于生化分析。使用BioAssay Systems的试剂盒测定甘油三酸酯和游离脂肪酸的水平。
原代人肝细胞
冷冻保存的原代人肝细胞得自Thermo Fisher Scientific(第一名捐助者),BioIVT(第二名捐助者)或Lonza(用于禁食研究)。我们的大多数实验都使用了赛默飞世尔科技(第一供体)的肝细胞,除非在本文其他地方进行了说明(第二供体或禁食研究)。为了体外培养原代人肝细胞,准备了细胞培养基,并按照制造商的建议遵循标准程序。对于GW 7647(Sigma)处理,首先将过夜接种的原代人肝细胞(无血清)在William Medium E中孵育4小时,然后进行对照(DMSO)或10μMGW 7647处理另外4小时。为了进行应激治疗,将过夜接种的原代人肝细胞(无血清)用5%胎牛血清或100μMH处理。2 O 2持续4 h。对于热休克处理,首先将细胞在42°C下培养30分钟,然后在收获前在37°C下恢复4 h。为了检测敲除LINC01018的基因表达,在铺板后4小时,将人原代肝细胞用sh-lacZ或sh1-LINC01018腺病毒以MOI为10感染48 h,然后从这些细胞中提取RNA,并进行Real-进行PCR以测定基因表达。
腺病毒生产和体内腺病毒管理
使用以下序列设计LINC01018,人类HuR和小鼠HuR的shRNA(LINC01018 shRNA1:CCTTAAACTTGTACCACTT; LINC01018shRNA2:GCAAGAAGACCCAGCTATT;人类HuR shRNA:GGCTTTGTGACCATGACAA;小鼠HuR shRNA:GGTTTGGGCGAATCATCAA)。发夹模板寡核苷酸由Integrated DNA Technologies合成,然后根据制造商的规程克隆到pAD / Block-it系统(Invitrogen)的腺病毒载体中。LINC01018的过表达构建体是通过使用人肝cDNA样本通过PCR扩增NR_024424.2的序列而产生的。随后将该序列克隆到pAdv5腺病毒载体中用于病毒包装。腺病毒在HEK293A细胞(Invitrogen)中扩增,并通过CsCl梯度离心纯化。纯化的病毒用PD10色谱柱(GE Healthcare Life Sciences)脱盐,并用Adeno-X快速滴定试剂盒(Clontech)滴定。腺病毒以5×10的速度静脉内注射到小鼠体内8 PFU /小鼠过表达,1-2×10 9 PFU /小鼠对撞倒实验。在双重击倒实验的情况下,首先混合两种效价相等的病毒,然后每只小鼠接受2×10 9 pfu总病毒。7天后,收集组织样品用于进一步分析。
RNA下拉和质谱分析
使用生物素RNA标记混合物和T7 RNA聚合酶(Ambion)在体外转录生物素标记的RNA,并使用RNeasy Mini Kit(Qiagen)在柱上消化DNA进行纯化。人源化的肝组织裂解物是通过在15到20冲程的dounce匀浆器中匀浆冰冻的肝组织而制成的,该匀浆器在补充了RNaseOUT,蛋白酶/磷酸酶抑制剂鸡尾酒,Panobinostat和Methylstat的ProteaPrep两性离子细胞裂解缓冲液中进行。首先根据制造商的说明制备BcMag单体抗生物素蛋白磁珠(Bioclone),然后立即在RNA捕获缓冲液(20 mM Tris-HCl [pH 7.5],1 M NaCl和1 mM中进行RNA(20μg)捕获。在室温下搅拌30分钟(EDTA)。用NT2缓冲液(50 mM Tris-HCl [pH 7.4],150 mM NaCl,2,和0.05%NP-40),并与在补充有50 U / ml RNaseOUT,2 mM二硫苏糖醇,30 mM EDTA和0.02 mg / ml肝素的NT2缓冲液中稀释的30 mg细胞裂解液在4°C旋转孵育2 h 。依次用NT2缓冲液(两次),含有500 mM NaCl的NT2高盐缓冲液(两次),含有1 M NaCl的NT2高盐缓冲液(一次),含有750 mM KSCN的NT2-KSCN缓冲液依次洗涤RNA结合蛋白复合物。 (两次)和PBS(一次)在4°C下放置5分钟,然后用PBS中的2 mM D-生物素洗脱。依次用10 mM Tris(2-羧乙基)膦盐酸盐还原样品(无RNA对照的一个样品和LINC01018下拉样品的一个样品),用20 mM氯乙酰胺烷基化,然后在37°C下用胰蛋白酶消化过夜。将所得的肽用ZipTip(Merck Millipore,MA)脱盐,然后真空干燥。然后将样品用0.1%甲酸复溶,并在基于Orbitrap Lumos(Thermo Fisher Scientific,加利福尼亚州圣何塞)的nanoLCMS系统上,以数据依赖采集(DDA)模式进行分析。肽段在50厘米长的EasySpray PepMap RSLC C18色谱柱(赛默飞世尔科学)上分离,线性梯度为5-30%的乙腈在40分钟内释放。首先在375–1500 Da质量范围内以120 k分辨率和4e5自动增益控制(AGC)对前体离子进行了测量。它们随后以30%能量的高能碰撞离解(HCD)进行破碎。并以30k分辨率和5e4 AGC记录了碎片光谱。使用Mascot(v2.6,Matrix Science Inc.,波士顿,马萨诸塞州)通过Proteome Discoverer 2.2平台(Thermo Fisher Scientific)针对SwissProt Human数据库搜索得到的LCMS数据。搜索参数分别是前体离子和碎片离子的10ppm和0.02 Da质量容差,两次胰蛋白酶解理以及作为固定修饰和蛋氨酸氧化变量的半胱氨酸氨基甲酰甲基化。蛋白质和多肽的错误发现率(FDR)截止值为1%。
RNA免疫沉淀(RIP)
为了制备肝组织裂解物,使用DIP匀浆器在RIP缓冲液(150 mM NaCl,20 mM Tris pH 7.4、1 mM EDTA,0.5%Triton X-100和蛋白酶/磷酸酶抑制剂鸡尾酒)中匀浆冷冻的肝组织,进行15-20次冲程匀浆。和RNaseOUT)。对于每个RIP,首先将10μg兔IgG或HuR -RIP验证抗体(MilliporeSigma)与30μl洗涤过的Dynabeads®Protein G在300μl补充了0.2 mg / ml BSA,0.2 mg / ml肝素和0.2 mg / ml RIP缓冲液中孵育ml EcoRI tRNA处理一小时。然后将抗体偶联的珠子添加到5 mg肝组织裂解物中,该裂解物在500μlRIP缓冲液中稀释,并在4°C轻轻旋转孵育3小时。将小珠用RIP缓冲液短暂洗涤五次。在最后一次洗涤中,将五分之一的珠子用于蛋白质分析,并将其余的珠子重悬于1 ml的Trizol中以进行RNA提取。分离出共沉淀的RNA,并通过RT-PCR进行分析。将表达水平与来自输入的表达水平进行标准化,然后将倍数富集与IgG对照进行比较。
芯片分析
根据制造商的规程,使用Simple ChIP酶染色质IP试剂盒(Cell Signaling Technology)进行冷冻肝组织的ChIP测定。使用RNA Pol II ChIP验证的抗体(MilliporeSigma,Cat:17-620)或以兔IgG作为阴性对照进行免疫沉淀。每个ChIP中的DNA使用人类特异性引物通过定量实时PCR分析确定,该引物扩增覆盖基因转录起始位点的基因组序列(补充数据 7)。通过将ChIP DNA的量归一化为输入DNA来计算RNA Pol II的相对富集,并将其与IgG对照作为倍富集进行比较。
RNA提取和定量实时PCR分析
使用Trizol试剂(Invitrogen)从肝脏组织或细胞中分离总RNA。在不使用Turbo DNA的DNase(Ambion)处理后,使用1μgRNA用SuperScript®III First-Strand Synthesis系统(Invitrogen)进行逆转录。在ViiA™7 Real-Time PCR系统(Applied Biosystems Inc.)上进行实时定量RT-PCR。PCR程序为:在95°C下2分钟30 s进行酶活化,在95°C上进行40个15 s循环C,在60°C下1分钟。进行熔解曲线分析以确认实时PCR产物。为了检测人源化肝脏样品中人基因的表达,设计了人特异性引物,并将定量相对于人16S rRNA水平进行了标准化。为了检测野生型小鼠中小鼠基因的表达,将18S rRNA用作内部对照。 7。
免疫印迹
为了进行免疫印迹分析,将细胞和组织溶解在含有磷酸酶抑制剂(Sigma)和蛋白酶抑制剂混合物(Roche)的1%SDS裂解缓冲液中。将裂解物进行SDS-PAGE,转移到聚偏二氟乙烯(PVDF)膜上,与一抗孵育,然后与荧光偶联的二抗(LI-COR)孵育。使用定量荧光成像系统(LI-COR)可视化结合的抗体。HuR抗体购自MilliporeSigma(目录:03-102,1:1000稀释用于免疫印迹)。
胞质/核分级
将人源化的肝组织在含蛋白酶/磷酸酶抑制剂鸡尾酒和RNaseOUT的核分离缓冲液(250 mM蔗糖,10 mM Tris-HCl,pH 7.4、10 mM KCL,1.5 mM MgCL2、1 mM EDTA,1 mM EGTA)中匀浆。将裂解物以1000× g离心 3分钟以沉淀出细胞核,并将上清液用作细胞溶质级分。在提取RNA之前,先用含0.1%NP-40的核分离缓冲液短暂洗涤核托盘。通过总量计算基因在细胞质和核级分中的相对表达,并将细胞质级分设置为1。
体外翻译分析
体外翻译分析使用TnT®快速偶联转录/翻译系统(Promega)进行。简而言之,通过PCR扩增,将T7启动子连接至LINC01018 DNA的5′末端。然后将纯化的PCR产物用作模板,按照制造商的规程进行体外翻译分析。使用生物素化的赖氨酸标记新合成的蛋白质,然后使用IRDye链霉亲和素(LI-COR)将其可视化。黄色荧光蛋白(YFP)的开放阅读框用作阳性对照。
293A细胞中的RNA稳定性分析
在标准条件下培养293A细胞。将人CYP4A11(NM_000778.4)和LINC01018(NR_024424.2)的全长克隆到pAdv5载体(Invitrogen)中,以在293A细胞中表达。空的pAdv5载体用作pAd-LINC01018的对照。表达人HuR(目录号:RC201562)和对照(目录号:PS100001)的载体购自Origene。转染后24 h,将293A细胞用0.5μg/ ml放线菌素D处理0、2和4 h。提取RNA并进行RT-PCR以确定每组中CYP4A11的相对表达(标准化至时间点0小时)。
报告摘要
有关研究设计的更多信息,请参见与本文链接的《 自然研究报告摘要》。
|
|