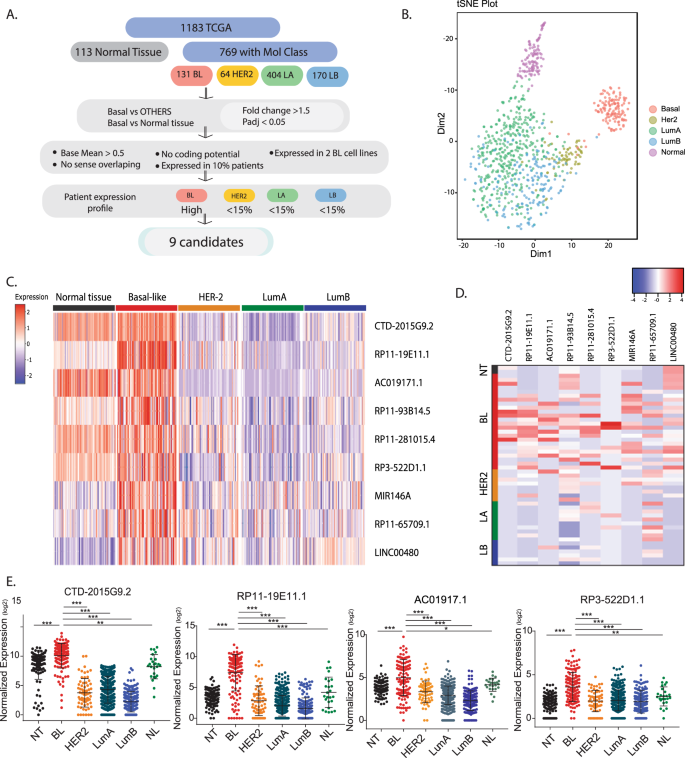
BL乳腺癌中过度表达的临床相关INcRNAs的鉴定 为了识别在BL乳腺癌中起作用的INcRNAs,我们使用了来自1183名患者的RNA测序(RNA-seq)数据,这些数据可以在癌症基因组图谱(TCGA)数据库中找到。我们用PAM 50分子亚型注释对肿瘤进行分类, 16 获得由131 BL,64 HER 2,404 LA和170 LB亚型所代表的769名患者的最终队列(图)。 1A )。为了进一步分析,我们排除了25个最初被归类为正常类(NL)亚型的肿瘤。我们和其他人以前已经证明,INcRNA的表达反映了乳腺癌的亚型特异性。 13 ,14 ,17 因此,对前500个在患者中表达的incRNAs的t-sne(t分布随机邻居嵌入)分析显示,这组患者的分子亚型聚类,类似于使用标记基因获得的聚类,即编码基因和非编码基因的混合(如图所示)。 1B ,补充图。 1A )。通过差异表达基因分析(DEseq分析),我们发现在BL亚型中,与正常组织和其他亚型相比,过度表达的INcRNAs有一个子集(>1.5倍的变化)。为了丰富临床相关的INcRNAs,我们筛选出那些在不到10%的患者中表达的低基线RNA(<0.5)。为了研究其在体外的功能,我们分析了一组细胞系的RNA-seq数据,并筛选出至少在两个BL乳腺癌细胞系中表达的INcRNAs。为了进一步限制抑癌基因在BL乳腺癌中的高表达,我们选择了在BL乳腺癌中高表达和在其他亚型中表达最少的基因(FPKM(每百万个外显子模型的片段数)<1)(图1)。 1C,d )。有趣的是,这份报告列出了九种候选基因,其表达情况足以通过t-SNE分析对BL亚型肿瘤进行聚类(补充图)。 1B
图1:利用tcga数据库中的患者数据鉴定基底样乳腺癌亚型的特异性incRNAs。 a 用于识别INcRNA候选的管道流程图。 b T-SNE图起源于500例患者中表达最强的INcRNA。 c 热图显示9种INcRNA候选基因在不同乳腺癌亚型中的表达。 d 热图显示九种候选细胞在一组按分子亚型分类的细胞系中的表达情况。 e 点图显示在乳腺癌亚型内的每个患者的表达水平,选择的INcRNA候选。平均±SD代表每种情况。* P < 0.05; **p < 0.01; ***p < 0.001 (ANOVA). NT normal tissue, BL basal-like, HER2 her2-enriched, LA luminal A, LB luminal B, NL normal-like.
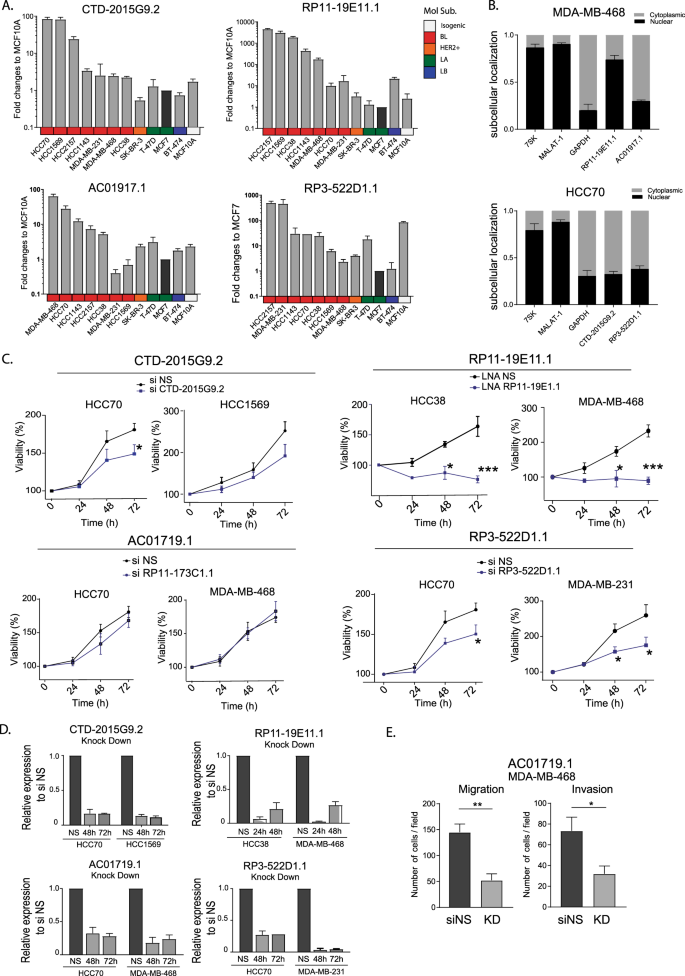
BL乳腺癌中过度表达INcRNAs的体外验证及功能筛选 为了研究这些INcRNA在肿瘤生长或肿瘤进展中的潜在作用,我们采用定量逆转录聚合酶链反应(qRT-PCR)技术分析了它们在代表乳腺癌不同分子亚型的细胞系中的水平。我们选择那些在至少两个BL细胞株中通过qRT-PCR和过表达(OE)检测到的incRNAs来进行进一步的研究,与其他癌症和/或正常乳腺细胞株相比,至少有10倍于此。INcRNAs CTD-2015G9.2、RP11-19E11.1、AC 01917.1和RP3-522D1.1符合这些标准,由于它们在细胞系中的表达模式与病人观察到的一致,因此被选择用于进一步的功能研究(图6)。 1E 和 2A )。接下来我们研究了这些incRNAs的定位,因为这可以提供对其潜在功能的洞察。4种候选基因中有3种主要表现为细胞质定位(类似于甘油醛3-磷酸脱氢酶(GAPDH)),RP11-19E11.1表现出较强的核定位,与核非编码RNA(NcRNA)对照、Malat-1和7SK的水平相似。(无花果) 2B 2 )。较高水平的INcRNA AC 01917.1的患者有更好的生存状况( p 值0.035),而RP11-19E11.1水平较高的患者生存效果较差( p 价值0.041)。然后,我们询问这些incRNAs是否在癌细胞的生长和迁移中起了作用。为了评估这一点,我们设计了小干扰RNA(SiRNAs)对细胞质INcRNA和锁定核酸(LNA)GapmeRs(LNAs)来降低核INcRNA。最初,我们用两种过高表达INcRNA候选基因的细胞株来测定转录敲除后的细胞活力(见图)。 2C,d )。值得注意的是,在所有缺失CTD-2015G9、RP11-19E11.1或RP3-522D1.1的细胞系中,存活率都降低了,但AC01917.1基因敲除后没有观察到任何影响。因此,我们测试了这种INcRNA在细胞迁移和/或侵袭中是否重要(图一)。 2E )并发现两种不同细胞株AC 01917.2在Boyden室迁移试验中的迁移和侵袭受到抑制。
图2:INcRNA候选基因的体外验证和功能筛选。 a 用qRT-PCR方法在一组细胞系中获得了4个候选细胞的RNA表达。以MCF 7细胞株为参照。 b 细胞核-细胞质分馏后用qRT-PCR方法检测INcRNA候选体的亚细胞定位。 c 在不同时间点对两种不同的细胞株进行抑制后(siRNA 20 nm或LNA 50 nm)的存活率(MTT)测定。 d 转染48和72h后,两个细胞系的INcRNA候选基因RNA水平下降。 e INcRNA AC 01719.1基因敲除后MDA-MB-468细胞的迁移和侵袭。实验至少进行三次,一式三份。数据为平均值±扫描电镜。* P < 0.05; **p < 0.01; ***p < 0.001 (two-tailed unpaired t 测试)。
因此,我们的研究表明,我们已经确定了一小群在患者和细胞系中都有BL亚型特异性表达的INcRNA候选基因。重要的是,我们在此基础上聚类BL肿瘤的能力表明,这些INcRNAs可能具有预后价值,并在肿瘤的发展和癌变中发挥潜在的作用。
BL特异性和染色质相关INcRNA RP11-19E11.1的遗传特性研究 在40%的BL患者中,细胞核INcRNA RP11-19E11.1高表达。我们发现,细胞活力在被击倒后会受到严重的损害,并且高水平的INcRNA患者的预后很差。因此,我们试图深入地描述它的潜在功能,并在我们剩余的研究中将重点放在这个INcRNA上。
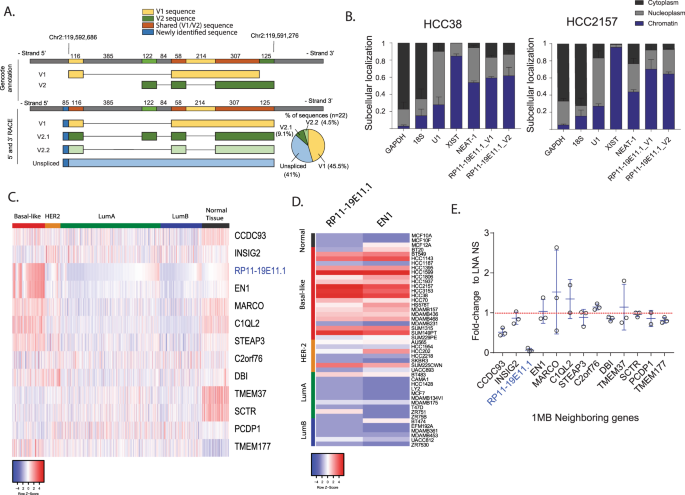
RP11-19E11.1是一种基因内的INcRNA。这个INcRNA的注释识别了两个不同5‘和3’末端的转录体(图)。 3A )。为了验证该INcRNA的真实5‘端和3’端,我们利用高表达INcRNA(HCC 2157)的细胞系的核分数进行了cDNA末端的5‘和3’快速扩增(RACE)。我们的结果表明,与以前的注释不同,变体1和2(V1和V2)具有相同的3‘端(图1)。 3A )。此外,我们无法检测到带注释的V2的5‘端的任何转录本,而具有与V2相同内含子的两个不同的异构体共享V1的5’端(图1)。 3A )。RACE克隆测序结果表明,所有变异体在V1注释的5‘端包含85个额外的核苷酸。
图3:INcRNA RP11-19E11.1转录特性。 a 三种主要变体的5‘和3’种族结果的方案。 b 核质分馏和染色质沉淀后,用qRT-PCR方法检测V1和V2的亚细胞定位。数据以平均值±扫描电镜表示。 c TCGA患者INcRNA RP11-19E11.1中1Mb相邻基因表达的热图研究。 d RP11-19E11.1和EN1在一组细胞系中表达的热图。 e 点图显示每个细胞系RP11-19E11.1相邻基因的表达水平。每个基因的平均值为±SD。表达低于检测水平的基因值未显示。实验至少进行三次,一式三份。
接下来,我们将探讨这种INcRNA是否与染色质有关,因为许多核INcRNA在染色质重塑或转录调控等功能中发挥着作用。 18 ,19 我们的结果清楚地表明染色质的结合,类似于整齐-1或Xist,用作V1和V2的对照(见图)。 3B )。核内干扰RNA可调控邻近基因的表达 顺式 。为了研究这一假设,我们绘制了一个热图,显示所有基因在incRNA周围的1 Mb窗口中的表达(图1)。 3C )。引人注目的是,我们观察到RP11-19E11.1和EN1的表达之间存在着近乎完美的相关性,EN1是一种定位于13 kb以外的神经特异性转录因子,与基底样乳腺癌有关。 20 在多个细胞系中也观察到了这种相关性(见图)。 三维空间 )。然而,在INcRNA被击倒后,我们没有观察到包括EN1在内的邻近基因的表达有明显的变化(如图1所示)。 3E )。此外,EN1的敲除并没有显著改变RP11-19E11.1的表达(补充图)。 3A )。我们还注意到,这两种转录本的OE并不是由于染色体上这一区域的扩增(CBioPortal)所致。由于RP11-19E11.1和EN1的启动子在CpG岛上都富集,我们推测观察到的协同OE可能是由于这两种转录体启动子中DNA甲基化的解除所致。利用tcga甲基化数据,我们发现三重阴性患者在该区域甲基化CpG岛的百分比明显低于其他癌症或正常组织亚型(补充图)。 3B )。DNA甲基化是哺乳动物基因组中必不可少的调控层,控制着包括发育在内的多种生物学过程。DNA甲基化模式在癌症中发生了深刻的改变,包括通过启动子高度甲基化抑制肿瘤抑制基因的转录,以及通过全球低甲基化激活基因转录。 21 我们的数据表明,RP11-19E11.1的表达可能由于含有这种INcRNA的一个大的染色体区的低甲基化而在基础肿瘤的子集中被解除管制。由于所鉴定的所有RP11-19E11.1转录本均显示核定位和染色质结合,因此我们认为这种RNA的功能与染色质相关过程的调控有关。
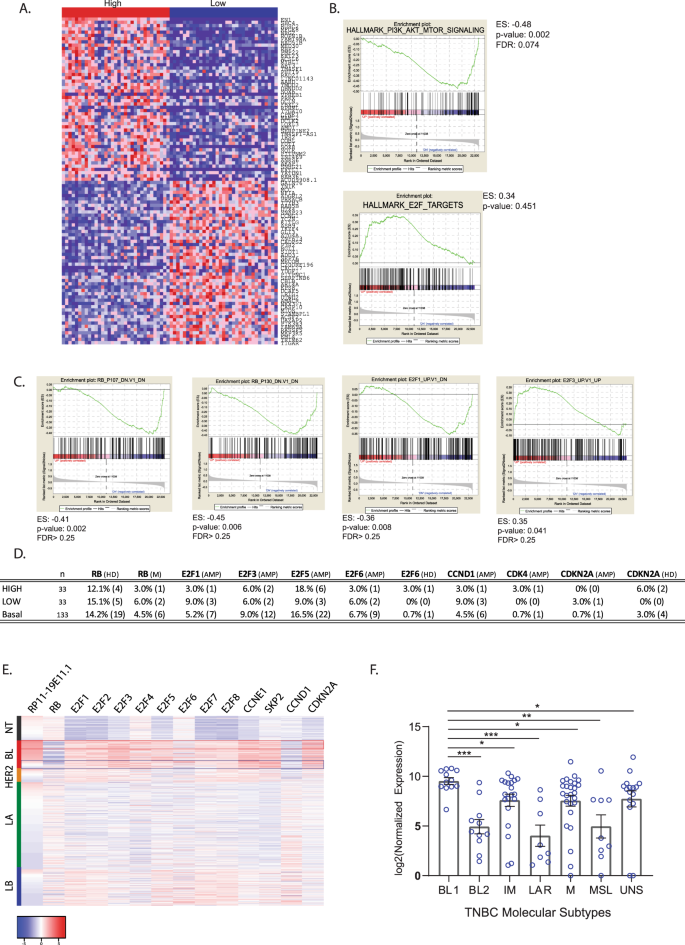
高rp11-19E11.1的患者表现出特殊的致癌信号。 由于RP11-19E11.1高水平患者的预后比低表达者差,我们询问过表达RP11-19E11.1的患者是否有特定的致癌信号。为此,我们比较了BL患者RP11-19E11.1高表达和低表达(1q对4q), n =33(每组)使用GSEA(如图所示)。 4A )。有趣的是,RP11-19E11.1水平较高的患者表现出明显的下调特征性磷脂酰肌醇-3-激酶/Akt/哺乳动物的雷帕霉素信号通路的靶点,并上调Rb/E2F的致癌通路(如图所示)。 4B,c )。然后,我们通过扩增、缺失或突变来评估这个轴的某些主要效应在癌症中是否有任何改变(Rb,E2F,CDKN2A,CCDN 1,CDK 4),这些改变可能与incRNA的表达模式有关。 22 (无花果) 4D )。解除对这一途径的管制是众所周知的在乳腺癌中普遍存在,并与不良结果有关。 23 我们没有发现高和低RP11-19E11.1的患者在任何可以激活这些通路的基因改变的发生率上有显着性差异(图一)。 4D,e )。由于RP11-19E11.1高表达的患者有一个特定的致癌信号,我们分析了这种INcRNA的表达是否被限制在一个独特的TNBC分子亚型上,正如另一个成熟的分类方案所定义的那样。 6 ,24 102例TNBC患者在这6种不同亚型中,BL1亚型中RP11-19E11.1明显富集(图1)。 4F
图4:基底样高发患者的GSEA分析 n =33比低( n =33)RP11-19E11.1的表达水平。 a RP11-19E11.1高、低患者上、下位基因热图。 b GSEA丰富了标记存储库的结果。 c GSEA的富集导致致癌通路的形成。 d 高发患者E2F轴基因改变 n =33)或低( n =33)RP11-19E11.1水平或基底样亚型(标记为基底层)。 e 乳腺癌亚型中E2F-和E2F调控基因的热图。 f 点图显示在102例TCGA(Mann-Whitney试验)TNBC患者中,Vanderbilt分类所鉴定的分子亚型中每个患者RP11-19E11.1的表达情况。给出了每种条件下的平均±扫描电镜。* P < 0.05; **p < 0.01; ***p < 0.001. HD homodeletion, M mutation, AMP amplification.
这些结果提示,除遗传背景外的其他因素可触发E2F的致癌信号,这一特征与BL乳腺癌预后不良有关。
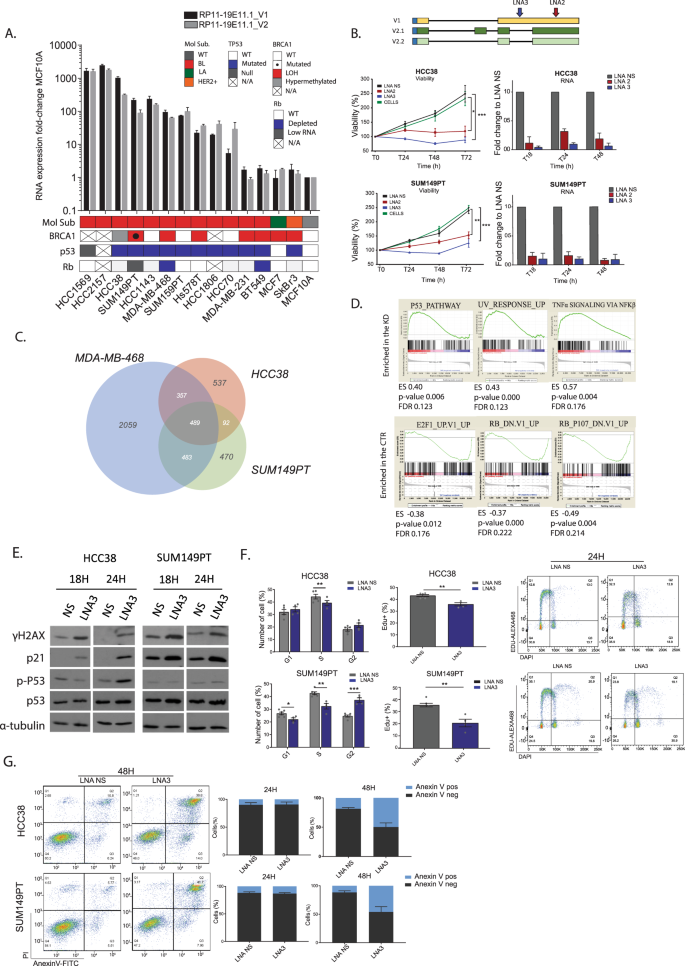
incRNA rp11-19E11.1基因敲除诱导细胞周期阻滞和凋亡 我们扩展了我们的分析小组的BL细胞株,以研究两种变体的INcRNA RP11-19E11.1的水平。我们还分别用两种不同的LNAs(MTT,3-(4-5-二甲基噻唑-2-基)-2,5-二苯基四氮唑溴)对两种不同的LNAs进行了活性测定,分别针对两种不同细胞株HCC 38(LNA 2和LNA NS在72h时的存活率)进行了活性测定。 p 值0.017,用于LNA 3和LNA NS p 值<0.001)和SUM149PT(LNA2vs LNA NS在72h存活时间) p 值0.002,用于LNA 2和LNA NS p 值<0.001)(如图所示)。 5A,b )。结果发现,转染后18 h,LNA 3的敲除效率最高(>90%,LNA 2>80%)。此外,我们观察到,无论使用哪种LNA,这两种变异体在被击倒后都会被下调,这表明它们可以针对无剪接的转录本(补充图)。 4 )。我们接下来在三个细胞系中进行了靶向RP11-19E11.1的LNAs转染后的RNA-seq。489个基因在三种细胞系中普遍被解除管制(折叠变化>2,假发现率<0.1,图)。 5C )。为了确定可能与INcRNA功能相关的主要途径,我们进行了GSEA分析,并对解除管制的基因中的致癌途径和转录因子基序的富集进行了评估。有趣的是,我们的分析表明E2F/Rb致癌通路和E2F转录因子基序丰富的基因在RP11-19E11.1敲除后丢失。 5D )。相反,表达上调的基因包括p53途径、紫外线(UV)反应和通过核因子-αB的肿瘤坏死因子-κ信号(图1)。 5D
图5:INcRNA RP11-19E11.1的功能特征。 a 用qRT-PCR方法检测基底样细胞系中V1和V2的RNA水平。以MC10A为参照。 b 用两种不同靶点的LNAs(LNA 2/LNA 3 25 NM)对HCC 38和SUM149PT细胞株的存活率(MTT)进行检测。 c 3种细胞系(LNA 1+LNA 2 50 NM)在24 h后,其基因普遍解除(折叠变化>2,假发现率(FDR)<0.1)的Venn图。 d 在INcRNA RP11-19E11.1基因敲除后,GSEA的富集导致标记和致癌途径的增加。 e γH2AX、P53、p-p53和p21在转染后18和24h(25 Nm)后的蛋白质分析。 f LNA3转染HCC 38和SUM149PT细胞24h后,细胞周期分析和EDU掺入分析(PI-Edu双染)。每个点代表一个度量。 g Annexin-V分析LNA3转染HCC 38和SUM149PT细胞后24h和48h后的变化。实验至少进行三次,一式三份。数据为平均值±扫描电镜。* P < 0.05; **p < 0.01; ***p < 0.001 (two-tailed unpaired t 测试)。
根据这些转录改变,我们对RP11-19E11.1基因敲除后的DNA损伤标记物(γH2AX)和p53途径蛋白进行了Westernblotting分析。我们观察到18h后γH2AX的大量诱导,P53水平在敲除后升高,与其典型靶标p21/CDKN1A(图1)一致。 5E )。上调其他典型的p53靶基因, GADD 45 和 PMAIP 1/Noxa ,在这两个细胞系中也观察到(补充图)。 5A )。因此,细胞周期分析显示,所有细胞系(HCC 38)的S期分数都会随之降低。 p 值0.017,SUM149PT p 值0.002),以及SUM149PT细胞的G2/M期块( p 5-乙炔-2‘-脱氧尿苷(Edu)掺入法(HCC 38)测定的dna合成抑制也证实了该值<0.001。 p 值0.0011,SUM149PT p 值0.0014)(如图所示。 5F )。鉴于p5 3靶基因的明显激活和DNA损伤反应,我们询问是否能在缺失这种INcRNA的细胞中诱导凋亡。事实上,用Annexin-V染色法进行凋亡分析显示,细胞在击倒后48小时内死亡(如图所示)。 5G )在这两个细胞系中。
这些观察表明,我们的GSEA分析与RP11-1E11.1基因敲除后所描述的表型密切相关,即E2F下调和细胞周期阻滞,然后激活凋亡通路。推测细胞凋亡可能与RP11-1911.1 E11.1基因敲除后的复制应激或DNA损伤反应有关。
p5 3下游基因的诱导是以一种部分不依赖p5 3的方式进行的。 p53基因突变在三重阴性乳腺癌中已被报道超过80%. 25 大多数突变位于DNA结合区,干扰p53与启动子的亲和力,改变转录靶基因的表达。 26 我们注意到p53缺失细胞系HCC 1569有较高水平的RP 11-1911.1,因此我们询问INcRNA的敲除是否能在p53靶基因中引起同样的反应。出乎意料的是,p21水平在这个细胞系中独立于p53被上调(补充图)。 c. )。P53突变的SUM149PT细胞系也得到了类似的结果。该细胞系p5 3的缺失在INcRNA敲除后并不能挽救细胞的存活,p21的水平在RNA和蛋白水平上均升高,而不依赖于p5 3蛋白水平(补充图)。 5B,d,e )。因此,凋亡诱导可以独立于p53功能,这与p53突变频率高的乳腺癌亚型有关。
incRNA rp11-19E11.1是E2F1的靶基因。 RP11-1E11.1高表达的患者有一个与激活E2F靶基因子集有关的特定基因标记。事实上,这种INcRNA的缺失会导致细胞周期阻滞,从而减少增殖,进而诱导凋亡基因。因此,我们研究了三种可能的方案,可以将这个INcRNA与E2F基因标记联系起来。首先,INcRNA可以控制E2F1的转录。第二,INcRNA可能受E2F转录家族成员的调控,因此在维持细胞增殖中起着不可或缺的作用;第三,INcRNA参与调控E2F下游基因的一个特定子集。第二和第三种可能性并不是相互排斥的。
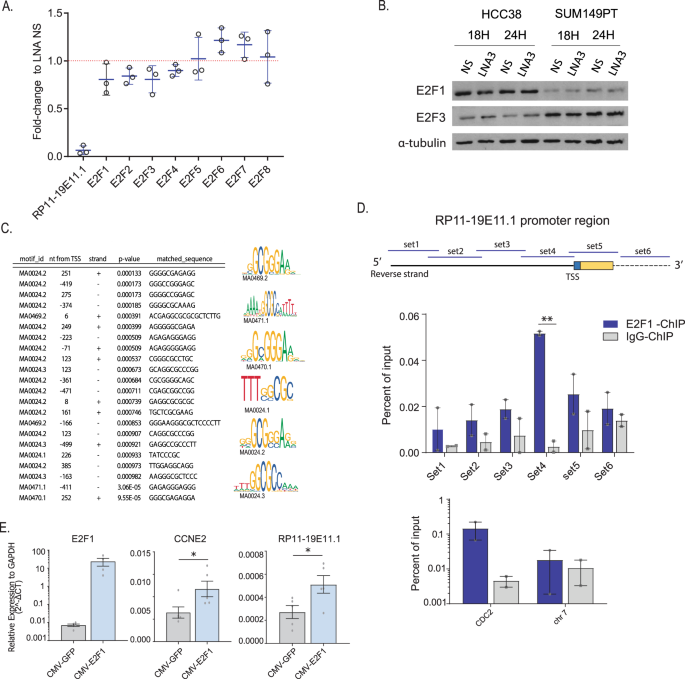
为了评估第一种选择,我们使用三个不同细胞株的rna-seq数据检测了RP11-1E11.1基因敲除后E2F家族的水平。结果没有显示任何E2F家族成员的RNA有明显的变化(图一)。 6A )。对E2F1和E2F3的蛋白水平也进行了评估,没有发现明显的变化(图一)。 6B )。因此,我们进一步评估了INcRNA作为E2F靶基因的可能性。启动子分析确定了一些潜在的E2F基序围绕着转录起始位点(TSS)。 p 值<0.001,图1。 6C )。为了验证E2F1与RP11-19E11.1启动子直接相互作用的假设,我们进行了染色质免疫沉淀(CHIP)分析。由于确定了几个可能的结合位点,我们设计了6个重叠引物集,它们跨越5‘种族鉴定的TSS的上游和下游区域。只有引物集4(−141+13)与对照相比明显富集。 p 值0.003)(如图所示。 6d )。同时,外源性E2F1的表达增强了RP11-19E11.1的表达。 p 值0.04),与另一个典型的E2F1目标基因CCNE 2( p 值0.017)(如图所示。 6E
图6:INcRNA RP11-19E11.1是E2F的靶基因。 a RP11-19E11.1基因敲除后E2F家族转录因子表达的折叠变化。每个点代表每个细胞株的折叠变化,每个基因的平均值±sd被表示。 b 用LNA 3对RP11-19E11.1后18、24h的E2F1和E2F3进行Westernblot分析。 c E2F转录因子RP11-19E11.1启动子区的基序富集分析。 d HCC 38中使用E2F1抗体的染色质免疫共沉淀(芯片)显示了两个生物副本(双尾未配对)。 t 测试)。 e 外源性高表达E2F1(双尾配对)后HCC 38和SUM149PT中CCNE 2和RP11-19E11.1的RNA水平 t 测试)。每个点代表一个度量。数据为平均值±扫描电镜。* P < 0.05; **p < 0.01; ***p < 0.001.
最后,关于RP11-19E11.1调控E2F家族靶基因的可能性,本研究表明RP11-19E11.1缺失后E2F基因信号在体外逆转,DNA损伤、DNA合成阻滞、凋亡通路激活。因此,我们不能排除某些E2F靶基因在RP11-19E11.1耗尽后细胞适应能力丧失而间接下调的作用。超出本报告范围的分析将有助于确定这种INcRNA在E2F转录因子家族直接或间接控制的基因调控中的作用。
E2F家族成员是众所周知的转录因子,控制细胞增殖的几个方面,从细胞周期进展到DNA损伤检查点和修复。已经确定了几个incRNAs为E2F靶,包括H19, 27 ANRIL 28 还有埃里克 29 这些基因在肿瘤细胞中均表现出异常表达,并在肿瘤的发生发展中发挥重要作用。我们认为RP11-19E11.1可能是这组INcRNA的另一个成员。
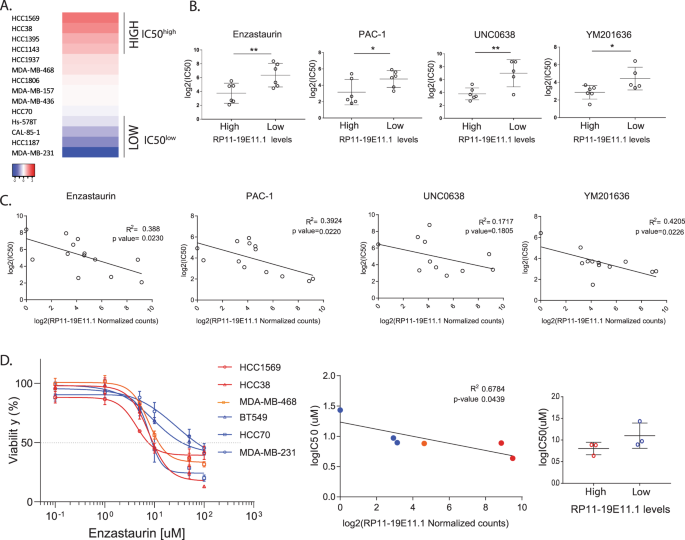
incRNA rp11-19E11.1表达对BL细胞药物敏感性的预测价值 高水平的INcRNA患者在BL亚型中显示出特定的基因特征。因此,我们询问BL亚型中的药物敏感性是否可以通过INcRNA RP11-19E11.1的表达水平来预测。为此,我们研究了不同表达水平的BL细胞系对来自肿瘤药物敏感性基因组学的267多个化合物的敏感性。 30 我们发现了14个BL细胞株的药物敏感性筛选数据,并将这些数据与从CCLE中获得的RNA-seq数据各自表达水平重叠。 31 根据rp11-19E11.1的表达情况,对4株上、4株底部细胞株进行排序,并比较其平均半数抑制浓度(IC)。 50 )高表达细胞株的平均IC值 50 (图1)最低的(见图)。 7A )。我们选择了那些显示IC的药物进行进一步的分析。 50 高 /IC 50 低层 比值<0.25,以确保各组间药物敏感性的生物学意义。对于那些显示IC的化合物 50 根据INcRNA的表达,比较6个上/下细胞系时,我们分析了药物敏感性与INcRNA表达水平之间是否存在相关性(图1)。 7b,c )。有趣的是,其中三个化合物的表达水平与敏感性呈显著负相关:pac-1( p 值0.022), p 价值0.023)和YM 201636( p 值0.023)(如图所示。 7C )。PAC-1是第一个开发出来的前caspase激活剂化合物,并于2016年被食品药品管理局指定为孤儿药物。 32 扎司他林是蛋白激酶C(PKC)的抑制剂,在治疗淋巴瘤的III期治疗中失败。 33 YM 201636是一种PIKfyve(磷脂酰肌醇磷酸激酶PIP5KIII)抑制剂,是一种参与PtdIns(3,5)P2生物合成的激酶。它的抑制破坏了被感染细胞的内膜运输和逆转录病毒释放。 34
图7:以INcRNA RP11-19E11.1水平作为预测值进行药物筛选。 a 根据RP11-19E11.1(来源于CCLE)的表达,可获得药物敏感数据的基底样细胞系排名。 31 b 识别出的敏感性显著不同的化合物(根据IC) 50 )比较RP11-19E11.1表达水平低或高的细胞系。 c RNA表达与药物敏感性的线性相关 50 ) b 。采用皮尔逊相关检验。不包括CAL-85-1细胞株(无表达数据). d 对苯司他林的结果进行了验证。数据为平均值±扫描电镜。* P < 0.05; **p < 0.01; ***p < 0.001 (unpaired t 除非有指明,否则进行测试。
为了验证筛选出的化合物,我们对RP11-19E11.1水平的qRT-PCR结果进行了排序,选出了3个上/下细胞系。采用与筛选方法相似的方法进行代谢试验的治疗时间和生活能力评估。我们发现无论是PAC-1还是YM 201636都没有观察到筛选细胞株的趋势(补充图)。 6a,b )。另一方面,除SUM149PT(补充图)外,Zenzastaurin在所有被测细胞系上都显示了预期的结果。 6C )。因此,我们决定包括另一个细胞株的RP11-19E11.1,MDA-MB-468的中等水平。在这里,我们复制了在筛选中观察到的结果,作为一个显著的负相关( p 在RP11-19E11.1水平与对扎司他林的敏感性之间观察到0.044)。 7D )。集成电路 50 在筛选中观察到的值有相同的趋势,但没有显着性。
这些结果是相当挑衅性的,因为pkc的活动导致了几条通路的激活,这些途径可以调节许多细胞过程,包括增殖和抗凋亡信号。 35 PKC通过直接磷酸化激活Raf1,进而导致Rb蛋白磷酸化,释放E2F转录因子。 36 此外,Raf1-RB蛋白相互作用的破坏导致肿瘤生长和血管生成抑制。 37 此外,PKC还能激活RAS,从而诱导E2F1的表达。 38 因此,PKC抑制可能是进一步探索E2F活化信号的基础乳腺癌治疗的一个有吸引力的靶点。