MTR4通过替代剪接促进癌症代谢转换,从而驱动肝脏肿瘤发生
MTR4通过替代剪接促进癌症代谢转换,从而驱动肝脏肿瘤发生
摘要
肿瘤发生需要从氧化磷酸化到糖酵解的代谢转换,以便为癌细胞提供能量和生物合成的底物。因此,阐明控制癌症代谢转换的机制很重要。MTR4是与核外泌体相关的RNA解旋酶,在RNA加工和监视中起关键作用。我们证明,MTR4经常在肝细胞癌(HCC)中过表达,并且是预测HCC患者预后不良的独立诊断标记。MTR4通过确保关键糖酵解基因(例如GLUT1和PKM2)的前mRNA的正确剪接来驱动癌症代谢。c-Myc与MTR4基因的启动子结合,对于HCC细胞中MTR4的表达很重要,表明MTR4是c-Myc在癌症代谢中功能的介体。这些发现揭示了MTR4在癌症代谢转换中的重要作用,并将MTR4作为治疗HCC的有希望的治疗靶标。
介绍
肝细胞癌(HCC)已成为世界上主要的癌症之一,治疗选择有限,死亡率不断提高1。与主要从氧化磷酸化(OXPHOS)获得能量的正常体细胞不同,人类癌细胞依靠糖酵解获得能量和生物合成2的底物。因此,阐明对于癌症代谢从OXPHOS转变为糖酵解的重要途径,将为癌症治疗提供更具体的治疗目标。尽管许多致癌途径(例如c-Myc途径)通过调节代谢基因的表达在促进癌症代谢转换中起重要作用3,各种类型的人类癌症表现出高水平的异质性,并且可能发展出独特的机制来诱导癌症的代谢重编程。例如,最近的研究表明,表达野生型p53的 HCC细胞依赖于p53-PUMA途径,通过PUMA介导的线粒体丙酮酸载体依赖性丙酮酸摄取破坏4来维持从OXPHOS到糖酵解的代谢转换。
基因表达的调节可以在许多水平上实现,包括转录和转录后机制。核外泌体使用募集特定RNA靶标进行加工的RNA结合辅因子复合物监测和降解RNA。两个主要的辅因子复合物,细胞质的Ski和核TRAMP复合物,参与募集RNA到外泌体5。RNA解旋酶(例如MTR4)与辅因子复合物结合并解开复合物RNA结构,以允许将RNA插入外来体中进行加工5。MTR4存在于TRMAP复合体,核外泌体靶向(NEXT)复合体和poly-A尾部外泌体靶向(PAXT)复合体中,用于RNA衰变,表明MTR4靶向广泛的RNA并调节其稳定性。MTR4尚未显示在选择性剪接(AS)中发挥重要作用,但外来体参与染色质重塑和各种的RNA如前mRNA剪接的正常处理6,7,8,9。尽管已经广泛研究了MTR4的生化功能,但仍不清楚MTR4在发育和疾病中的生理作用。
最近的研究表明,各种类型的人类癌症都可能发展出异常的AS景观,从而促成致瘤过程10。人类癌细胞中这些与癌症有关的AS模式可能是前mRNA剪接位点的突变和调控元件的变化或剪接体成分的改变11。例如,关键代谢基因(如PKM1 / PKM2)的AS事件的异常调节可能会导致癌症代谢重编程12。因此,为了提高当前治疗性干预措施的特异性和有效性,阐明异常RNA剪接的机制很重要,以便确定抑制癌症相关剪接事件的新靶标。聚嘧啶束结合蛋白1(PTBP1)是AS 13的重要调节剂。PTBP1与前mRNA的结合抑制了结合位点附近的外显子的剪接。但是,PTBP1与前mRNA的结合位点的序列是保守的,不能解释癌细胞中异常的剪接事件。因此,重要的是鉴定癌细胞中的PTBP1相互作用因子,这些因子可以调节pre-mRNA与PTBP1之间的相互作用,从而导致异常的癌症特异性剪接事件。
为了鉴定可能对异常的AS和HCC的肿瘤发生重要的蛋白质,我们分析了从基因表达综合(GEO)数据库(http://www.geo.org/en/)中的225个HCC组织和220个非癌性肝组织组装而成的转录数据集。(ncbi.nlm.nih.gov/geo/),发现在肝癌组织中RNA解旋酶MTR4的mRNA水平增加了(图 1a,b)。我们通过将PTBP1募集到其靶前mRNA来调节HCC相关的AS事件,证明了MTR4在促进HCC肿瘤发生和癌症代谢重编程中的重要作用。我们的发现揭示了肝癌中异常AS事件和癌症代谢重编程的潜在机制,并通过抑制HCC相关AS提供了治疗HCC的新治疗靶点。
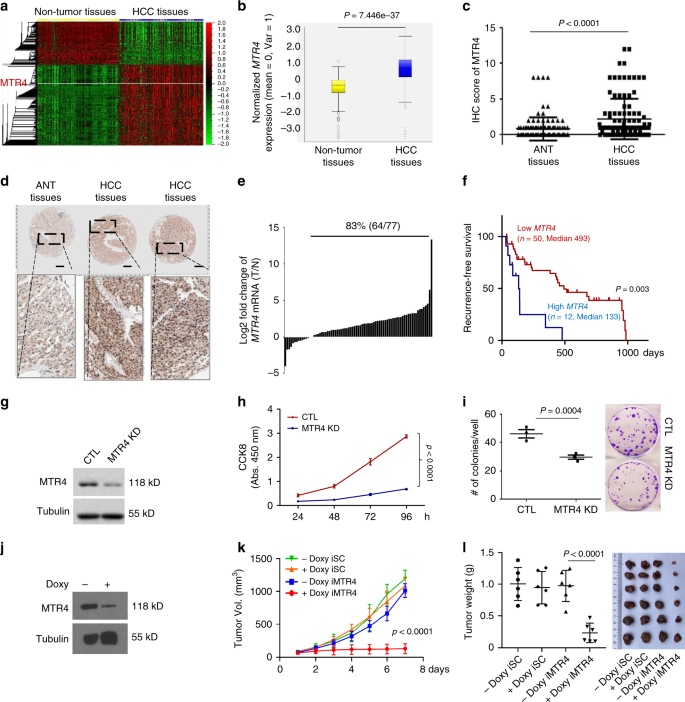
GSE14520数据集中非肿瘤组织(n = 220)和肝细胞癌(HCC)组织(n = 225)中全局mRNA表达谱的热图。MTR4的水平用白线表示。b箱线示出的相对mRNA水平MTR4 HCC组织(Ñ = 225)和非肿瘤组织(Ñ = 220)在GSE14520(GPL3921)数据集。差异的显着性通过两尾未配对t检验进行评估。指示p值。中心是框内的中位数,框的边界跨四分位间距,并且晶须可视化了5%和95%的数据点。C,d HCC样本(码片Ñ = 108)与相邻非肿瘤(ANT)样品(Ñ = 108)用抗MTR4抗体染色和染色的强度进行扫描,并取得。显示了代表性的免疫组织化学(IHC)图像。两尾不成对t检验。数据以平均值±sd p值表示。比例尺= 200 µm。e将HCC样本(n = 77)中的MTR4 mRNA水平与相应的ANT样本(n = 77)进行比较。83%(64/77)的HCC样品的MTR4 mRNA水平高于ANT。˚F的MTR4水平与肝癌患者的术后无复发生存率(RFS)呈负相关。患者RFS高MTR4 mRNA水平(Ñ = 12)是比低显著下MTR4(Ñ = 50)。生存率差异通过对数秩检验(Mantel Cox)进行评估。指示p值。g通过Western印迹证实了HCC细胞PLC / PRF / 5中MTR4的敲低。CTL,表达加扰shRNA的HCC细胞。微管蛋白被用作内部对照。从两个独立的实验中获得了一致的数据。h用CCK8分析法分析MTR4 KD和对照细胞的增殖。ñ = 3个生物学独立的实验。两组之间的差异通过双向方差分析计算,然后进行Bonferroni后测。数据以平均值±sd p值表示。我的控制和MTR4 KD细胞的集落形成试验。两组之间的差异通过两尾不成对t检验计算。数据以平均值±sd p值表示。n = 3个生物学独立的样本。j用1 µg / ml强力霉素(Doxy)处理4天后,通过蛋白质印迹法证实PLC / PRF / 5细胞(iMTR4细胞)中MTR4的诱导敲低。从两个独立的实验中获得了一致的数据。ķ,升的体积(ķ)和重量(升由PLC / PRF / 5细胞中表达在NSG小鼠加扰的shRNA(ISC)或iMTR4细胞形成的肿瘤的)强力霉素的每日腹膜内注射(20毫克公斤后测量-1体重)或模拟治疗8天。脱氧治疗后每天测量个体肿瘤体积。反复进行两次方差分析,然后进行土耳其的后期测试。指示p值。重复进行两次方差分析,然后进行Bonferroni后测。数据以平均值±sd p值表示。ñ 每组= 6个独立样本。在治疗结束时,比较每组中所有肿瘤的重量。曼·惠特尼检验。指示p值。 每组n = 6个独立样本。源数据作为源数据文件提供。
结果
HTR细胞的肿瘤发生需要MTR4
与mRNA表达数据一致,在108对配对的HCC组织和邻近的非肿瘤(ANT)组织中对MTR4进行免疫组织化学分析表明,HCC样品中MTR4的蛋白质水平显着高于ANT组织(图 1c,d,补充图 1)。为了揭示肝癌中MTR4表达水平的临床相关性,我们分析了切除的肝癌组织中MTR4表达水平与相应肝癌患者预后之间的相关性,表明肝癌组织中MTR4的表达水平呈负相关。肝癌患者的无复发生存期(RFS)(图 1e,f)。单因素分析表明,MTR4的高表达水平与HCC的四种病理学标志物显着相关,例如肿瘤大小(p = 0.027),肿瘤数目(p = 0.006),MVI(p <0.001)和分化(p = 0.043)(表 1)。用多变量Cox比例风险模型进行的分析表明,在调整肿瘤大小,肿瘤数量,MVI和分化后,MTR4的表达水平是肝癌RFS的独立预后标志物(表 1)。
为了研究MTR4在HCC肿瘤发生中的作用,我们沉默了MTR4在两种具有不同shRNA的HCC细胞系PLC / PRF / 5细胞和HepG2细胞中的表达(图 1g,补充图 2)。MTR4的敲低显著降低了增殖和肝癌细胞的集落形成能力(图 1H,我,补充图 2 B,C)。在已经建立的在免疫缺陷小鼠NODSCID HCC肿瘤MTR4的敲低诱导大大抑制肿瘤的生长(图 1J-1 ,补充图 2 E,F)。相反,在给定剂量下的强力霉素(Doxy)对在NODSCID小鼠中诱导性表达加扰shRNA的HCC细胞的肿瘤生长几乎没有影响(图 1k,1)。这些数据表明,MTR4是肝癌细胞发生肿瘤所必需的。
需要MTR4来维持HCC细胞的糖酵解
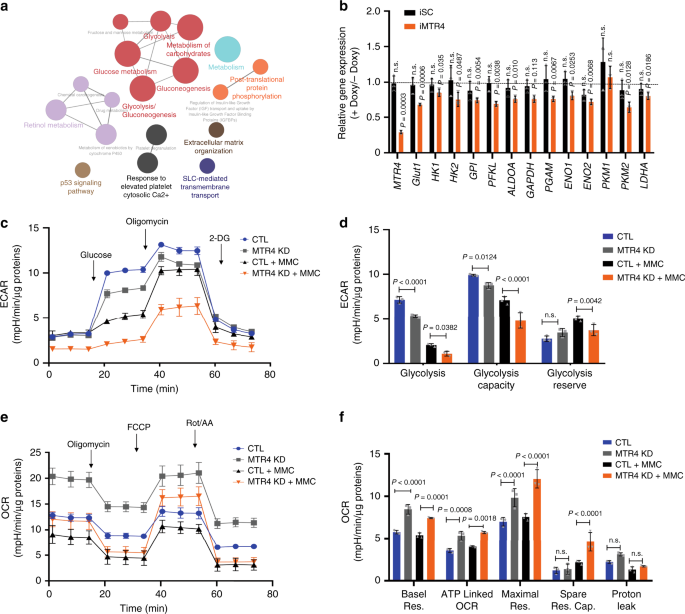
为了揭示HCC细胞依赖MTR4的肿瘤发生机制,我们在诱导MTR4敲除之前和之后对HCC细胞进行了RNA序列分析,以鉴定受MTR4影响的基因。通过MTR4诱导的敲除,等基因对照和HCC细胞之间超过1249个基因差异表达(倍数变化> 2.0)。基于KEGG功能注释和基因本体论(GO)术语,差异表达基因(DEG)显着丰富了代谢途径(补充图3)。使用共识路径数据库(CPDB),DEG丰富了信号传导和代谢途径,尤其是糖酵解和碳水化合物代谢14(图 2a)。基因集富集分析(GSEA)进一步揭示了在MTR4缺失的HCC细胞中糖酵解途径显着失调(补充图 3)。在这种情况下,敲除MTR4后,HCC细胞中几种关键糖酵解基因(例如GLUT1和PKM2)的表达降低(图 2b)。强力霉素不影响可诱导表达加扰shRNA的HCC中这些糖酵解基因的表达,表明仅使用剂量的强力霉素不会影响代谢基因的表达(图 2b)。因此,MTR4对于维持肝癌细胞中糖酵解基因的表达很重要。
在Doxy处理之前和之后对iMTR4细胞进行的全局基因表达分析确定了差异表达基因(DEG),这些基因通过途径分析富集于各种途径中,并可视化为重叠图。b在Doxy处理之前和之后,iSC和iMTR4细胞中糖酵解基因的mRNA水平。两尾配对t检验。数据以平均值±sd p值表示。ns,无关紧要。n = 3个生物学独立的样本。c,d以葡萄糖(10 mM),寡霉素(1μM)和2-DG响应的MMC(5 µg / ml)或DMSO作为溶剂对照的对照细胞和MTR4 KD细胞的细胞外酸化率(ECAR) (50毫米)。n = 3个独立实验。数据表示为平均值±sd,采用Tukey多重比较测试的双向ANOVA。指示p值。e,f响应于寡霉素(1μM),氟羰基氰化物苯hydr(FCCP,1.5μM),鱼藤酮/抗霉素A(腐烂)的对照细胞和MMC或DMSO作为溶剂对照的MTR4 KD细胞的耗氧率(OCR) /AA,0.5μM)。 每组n = 3个独立实验。数据表示为平均值±sd,采用Tukey多重比较测试的双向ANOVA。指示p值。呼吸,呼吸;上限,能力。源数据作为源数据文件提供。
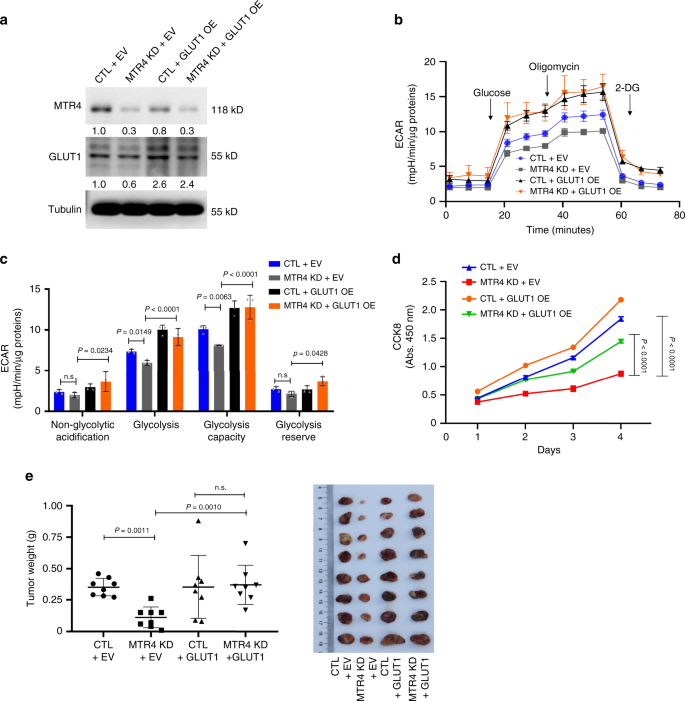
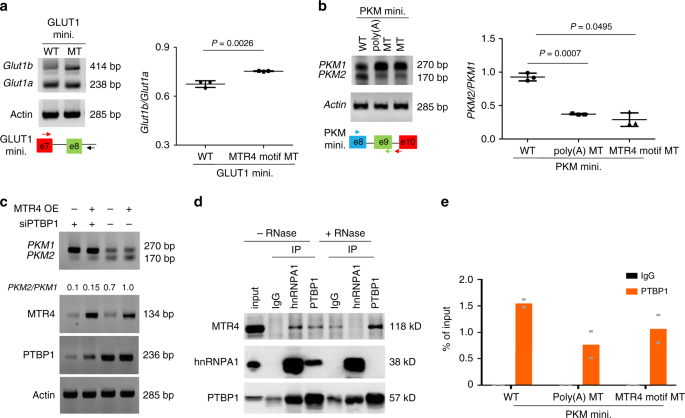
由于MTR4敲低会降低HCC细胞的增殖,因此为了评估MTR4敲低对代谢的影响,我们通过用丝裂霉素C处理细胞2小时来同步MTR4敲低和对照HCC细胞的增殖状态(补充图 4)。与MTR4敲低减少糖酵解基因表达的发现一致,MTR4表达的沉默降低了糖酵解活性并增加了氧化磷酸化,表明MTR4驱动了代谢从氧化磷酸化转变为糖酵解(图 2c-f)。敲除MTR4可减少关键糖酵解基因(例如GLUT1和PKM2)的表达(图 3a,b)。为了了解MTR4驱动的糖酵解和肿瘤发生的潜在机制,我们测试了MTR4依赖性糖酵解基因的表达对于介导MTR4驱动的肿瘤发生的重要假设。考虑到GLUT1在癌症代谢中的重要性,我们通过异位表达GLUT1基因拯救了MTR4沉默的HCC细胞中GLUT1的表达水平。MTR4沉默的HCC细胞中GLUT1蛋白水平的恢复部分挽救了体外糖酵解和细胞增殖以及体内肿瘤生长的缺陷(图 3b-e,补充图 5)。因此,依赖MTR4的糖酵解基因表达在驱动HCC的癌症代谢和肿瘤发生中起重要作用。
通过蛋白质印迹分析了对照和MTR4 KD细胞中GLUT1 的异位表达,以确定MTR4和GLUT1。显示了来自两个独立实验的代表性数据。b,c响应葡萄糖,寡霉素和2-DG的空细胞(EV)或表达GLUT1(GLUT1 OE)的质粒转染的对照细胞和MTR4 KD细胞中的ECAR。数据表示为平均值±sd,采用Tukey多重比较测试的双向ANOVA。 每组n = 3个独立实验。d用CCK8分析法分析指示细胞的增殖。数据表示为平均值±sd两次方差分析,采用Tukey多重比较测试。n = 3个独立的生物样本。e测量并比较了NSG小鼠中由指示细胞形成的肿瘤的重量。数据以平均值±sd的单向方差分析表示,然后进行Bonferroni后测。 每组n = 8个独立的生物样本。指示p值。源数据作为源数据文件提供。
MTR4调节HCC细胞中的选择性剪接(AS)
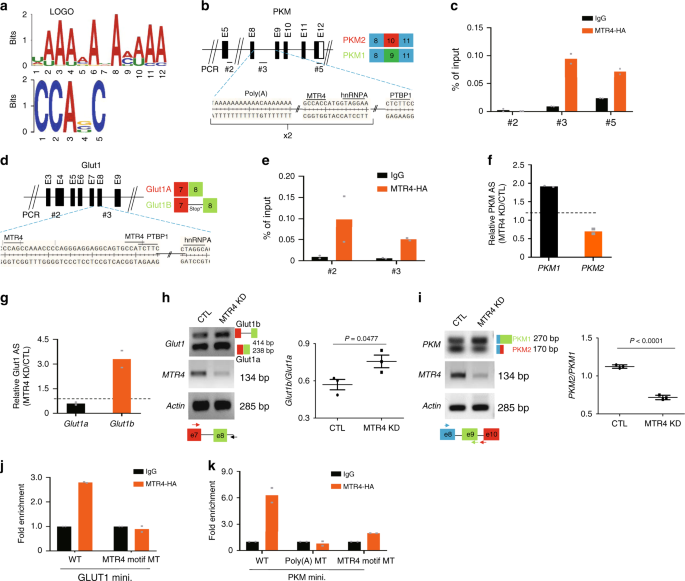
为了了解MTR4如何维持糖酵解基因的表达,我们进行了RNA免疫沉淀,然后测序(RIP-seq),以鉴定HTR细胞中MTR4结合的前mRNA。MTR4结合一半的转录物的内含子区域(1619/3301),表明pre-mRNA是MTR4结合的主要靶标(补充图 6a)。与RNA-SEQ数据一致,KEGG-功能注释分析表明,通过结合MTR4转录物在代谢和细胞周期途径显著富集的(补充图 6 B,C)。使用基元分析工具(例如MEME-Chip)筛选假定的MTR4结合基元15,已鉴定出两个MTR4结合基序:(UAAAAAA(U)AA(C)A(U)AA),考虑到MTR4是解旋酶解链的poly-A尾序列9的预期基序,以及基元CCAG(C)(U在pre-mRNA的内含子区域中发现的C)(图 4a)。
一个 MTR4结合基序,UAAAAAA(U)AA(C)A(U)AA(这是常规MTR4结合区,多聚(A)序列)和CCAG(C / U / G)C,通过的分析序列来定义使用主题分析工具MEME-Chip从RIP-seq获得3301个峰。b,d展示了MTR4和两个RNA选择性剪接因子PTBP1和hnRNPA的潜在结合基序的PKM基因(b)或GLUT1基因(d)的示意图。数字表示用于PCR的引物的位置。c,e RIP分析MTR4与PKM基因(c)或GLUT1基因的预测MTR4结合基序的结合。e由于缺少可用于免疫沉淀的抗MTR4抗体,因此将表达HA标记的MTR4的PLC / PRF5细胞与抗HA抗体或IgG进行免疫沉淀。通过RT-qPCR分析洗脱的RNA,并显示为输入的%。n = 2个独立实验。数据表示为平均值。数字表示PCR的引物位置(f,g)MTR4 KD细胞相对于对照细胞中PKM2 / PKM1(f)或GLUT1b / GLUT1a(g)的相对mRNA水平。虚线表示1。数据表示为平均值。n = 2个独立实验。^ h,我在MTR4 KD或对照细胞中,含有外显子的PKM(h)或GLUT1(i)基因组DNA的小基因(mini。)的小基因(mini。)的选择性剪接(AS)。用于PCR分析的引物用箭头指示。数据表示为平均值±sd两尾不成对t检验。指示p值。n = 3个独立实验。j,k RIP分析MTR4结合WT和MTR4结合基序突变PKM基因(j)或GLUT1基因(k)。将表达HA标签的MTR4的PLC / PRF5细胞与表达指示的小基因的载体结合,用抗HA抗体或IgG进行免疫沉淀。通过RT-qPCR分析洗脱的RNA,并显示为IgG的倍数变化。数据表示为平均值。n = 2个独立实验。源数据作为源数据文件提供。
虽然以前的研究已经表明,MTR4起着RNA监视和RNA稳定的重要作用,8,16,我们推测MTR4也可以通过调节前体mRNA的可变剪接调控mRNA水平。为了验证这一假设,我们分析了MTR4耗尽前后HCC细胞中的整体mRNA剪接。我们确定了与所有五个基本类型的AS模式相对应的两个样本组(KD与对照)之间的差异性可变剪接(AS)事件,从而导致1713个跳过的外显子(SE),131个备用5'剪接位点(A5SS),192个备用3'剪接位点(A3SS),307个互斥外显子(MXE)和103个保留了内含子(RI)差异AS事件(补充图6f)。我们使用了DAVID(https://david.ncifcrf.gov/home.jsp,版本6.8)对已识别的基因进行REACTOME途径分析,表明差异AS基因在代谢途径中富集(补充图 6c)。为了评估MTR4诱导患者HCC样本中异常AS事件的潜在意义,我们比较了MTR4 KD后MTR4的潜在靶基因与HCC细胞中经历异常AS的基因。我们的分析表明,预测的MTR4靶基因与MTR4 KD后HCC细胞中AS发生变化的基因之间存在显着重叠,这表明HCC中MTR4的过表达在诱导10例HCC患者的AS异常中起关键作用(补充图。 6d,e)。
MTR4通过将PTBP1募集到其目标前mRNA来调节AS
为了阐明MTR4如何调节HCC细胞中AS的机制,我们集中于关键的糖酵解基因GLUT1和PKM2,它们都是MTR4靶标,通过其前mRNA中MTR4结合基序的存在和标准RIP分析证实了这一点, MTR4与这些基因的前mRNA的内含子区域结合(图 4b–e)。后MTR4击倒,这些基因的mRNA前体的AS受到影响,使得mRNA的水平编码功能性GLUT1和PKM2被减小(图 4F,G)。相反,交替剪接的mRNA亚型GLUT1b,新型剪接的GLUT1 mRNA或PKM1的水平,增加(图 4f,g)。
为了确定MTR4是否通过预测的结合基序与pre-mRNA结合,我们构建了GLUT1和PKM2基因组DNA的小基因,该基因包含内含子与预测的MTR4结合位点侧接的外显子。与来自内源基因的数据一致,MTR4的敲低增加了从小基因转录的前mRNA与GLUT1b和PKM1亚型的剪接,表明这些小基因可以作为研究MTR4调控AS机制的模型(图 4h)。在这种情况下,小基因中预测的MTR4结合基序的突变消除了MTR4与从这些小基因转录的pre-mRNA的结合,从而确认MTR4结合了pre-mRNA的预测的结合基序(图 4j,k)。此外,类似于敲除MTR4的影响,小基因中MTR4结合基序的突变导致从小基因转录的pre-mRNA 分别与GLUT1b和PKM1同种型的剪接增加,表明MTR4与其靶标结合pre-mRNA是其在AS中的功能所必需的(图 5a,b)。
WT 的 AS和两个MTR4结合基序突变体(MT),包括PLC / PRF5细胞中GLUT1小基因中的poly(A)MT 。数据表示为平均值±sd两尾不成对t检验。指示p值。n = 3个独立实验。b WT的AS和两个MTR4结合基序突变体(MT),包括PLC / PRF5细胞中PKM小基因中的poly(A)MT 。数据以Dunnett多重比较测试的平均值±sd单向方差分析表示。指示p值。ns,无关紧要。n = 3个独立实验。C在有或没有MTR4过表达的PLC / PRF / 5细胞中,PTBP1敲低对PKM小基因AS的影响。使用(b)中所述的引物分析PKM1和PKM2同工型的水平。显示了来自两个独立实验的代表性数据。d在不存在或不存在RNase的情况下,PLC / PRF / 5细胞中内源性MTR4,hnRNPA1和PTBP1的共免疫沉淀分析。使细胞裂解物与所示抗体进行免疫沉淀(IP)。使用指定的抗体通过蛋白质印迹法检测免疫沉淀物中各种蛋白质的存在。由于IP级抗MTR4抗体不是市售的,因此我们没有进行MTR4 IP。显示了来自两个独立实验的代表性数据。ËRIP分析PTBP1与PLC / PRF5细胞中WT和MTR4结合基序突变体PKM小基因的结合。数据表示为平均值。n = 2个独立实验。源数据作为源数据文件提供。
为了了解MTR4如何调节AS事件,我们检查了MTR4和PTBP1之间的功能关系,后者是AS 13的关键调节器。尽管MTR4的过表达诱导了从PKM小基因转录的pre-mRNA剪接至HCC细胞中的PKM2亚型,但PTBP1在HCC细胞中的沉默抑制了从PKM小基因转录成PKM2的pre-mRNA的AS ,表明PTBP1参与其中在依赖于MTR4的剪接事件中(图 5c)。为了支持这一观点,共同免疫沉淀实验表明MTR4以不依赖RNA的方式与PTBP1相互作用(图 5d)。相反,MTR4以依赖于RNA的方式与另一种RNA结合蛋白hnRNPA1相互作用(图 5d)。因此,MTR4可能通过蛋白质间相互作用将PTBP1募集到其目标前mRNA。为了进一步支持这一观点,PKM小基因中MTR4结合基序的突变显着降低了PTBP1与从小基因转录的pre-mRNA的结合(图 5e)。总之,这些数据表明,MTR4通过将PTBP1募集到靶标前mRNA来调节其靶标基因的AS。
c-Myc直接激活癌细胞中MTR4的转录
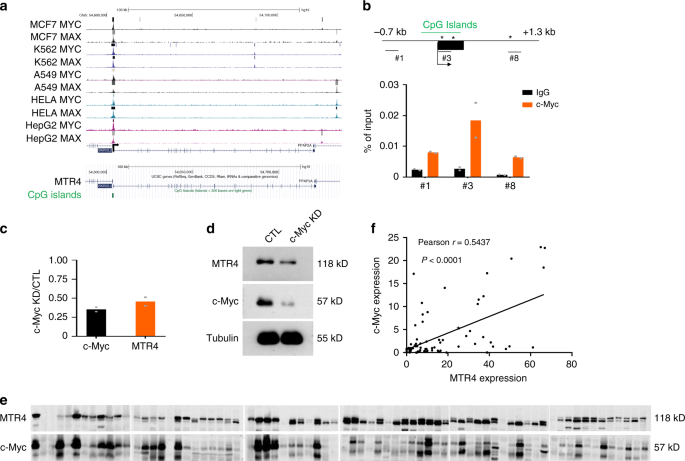
为了进一步了解MTR4途径在肝癌发生中的重要性,我们寻找了可能激活MTR4途径的上游调节剂。因为临界癌蛋白c-Myc的促进通过调节AS的关键糖酵解基因如PKM2部分糖酵解12,17,我们研究了c-Myc和MTR4之间的功能性相互作用。在ENCODE数据库中对几种癌细胞中c-Myc的染色质免疫沉淀测序(ChIP-seq)数据的分析显示,c-Myc与MTR4基因的启动子结合(图 6a)。CHIP分析证实了这一预测,确定了TSS附近的一个695 bp CpG Isalnds和两个可能被c-Myc结合的非经典E框(CACGCG,CACGAG)(图 6b))。敲除HCC细胞中c-Myc会降低MTR4的mRNA和蛋白水平的表达,这表明c-Myc直接激活MTR4的表达(图 6c,d)。该发现的生理相关性进一步得到以下发现的支持:c-Myc的蛋白质水平与患者HCC组织中MTR4的水平呈正相关(图 6e,f)。总之,MTR4是c-Myc在促进HCC癌症代谢中的关键下游介质。
a对ENCODE数据库中可用的人类癌细胞系中c-Myc的Chip-seq数据进行的分析预测,c-Myc与MTR4的启动子区域结合。b ChIP分析c-Myc与MTR4启动子内预测的c-Myc结合位点的结合。显示了MTR4调节区的示意图(距离TSS为-0.7到+1.3 kb)。箭头和黑色矩形分别代表转录起始位点(TSS)和外显子1。*表示规范的E框。绿条表示CpG群岛。数字表示PCR的区域。染色质DNA片段用抗c-Myc抗体或IgG进行免疫沉淀。数据表示为平均值。n = 2个独立实验。Ç的mRNA水平的c-Myc和通过q-RT-PCR检测MTR4,并通过肌动蛋白标准化。数据表示为平均值。n = 2个生物学独立的样本。d敲除HCC细胞中c-Myc后c-Myc和MTR4的蛋白质水平。显示了来自两个独立实验的代表性数据。e,f通过Western blotting(e)分析HCC患者样品中c-Myc和MTR4的蛋白水平,并分析其相关性(f)。蛋白质样品在多种凝胶上电泳,一个普通样品在所有凝胶上电泳,作为定量对照。两尾Pearson相关检验。p值与皮尔逊相关系数r被指示。n = 68个HCC患者样本。源数据作为源数据文件提供。
讨论区
癌细胞的遗传异常和表观遗传异常均有助于癌症进展和耐药性。越来越多的数据表明,癌细胞会出现异常的AS概况,从而显着增加致癌网络促进肿瘤发生的复杂性,但同时提供潜在的癌症特异性治疗靶点11。因此,确定诱导人类癌症中异常AS事件的致癌途径至关重要。我们发现,通过维持HCC细胞中关键糖酵解基因的表达,RNA解旋酶MTR4是HCC肿瘤发生所必需的。MTR4通过调节大量前mRNA(包括从关键糖酵解基因转录的前mRNA)的AS来实现这种代谢作用。由于MTR4在包括HCC在内的几种类型的人类癌症中过表达,因此MTR4的目标pre-mRNA与HCC患者样品中经历异常AS的pre-mRNA显着重叠10,MTR4可能是HCC致癌性AS事件的主要调节者。连同我们的发现,即已建立的HCC中MTR4的急性耗竭消除了体内肿瘤的生长,我们建议MTR4可以代表治疗HCC的有效且特异性的治疗靶标。
作为一种RNA解旋酶,已经进行了广泛的研究来阐明MTR4在RNA加工中的生化作用,但是对MTR4在生理学和肿瘤发生中的作用仍然知之甚少。先前的研究表明,MTR4的主要作用包括与RNA子集(例如病毒RNA)的3'UTR结合,并将其靶向降解,因此参与调节RNA的稳定性18。我们的发现揭示了MTR4在AS中的新作用,AS是一种高度调节的RNA加工活性,对于肿瘤的发生非常重要。在这种情况下,MTR4通过与前mRNA的内含子区域结合并通过蛋白质与蛋白质的相互作用将PTBP1募集到其目标前mRNA来调节AS。MTR4靶向的pre-mRNA富含代谢和细胞周期途径,表明MTR4具有多样化和广泛的生理和致癌功能。为了支持该观点,我们证明了糖酵解基因的MTR4依赖性表达对于HCC细胞的糖酵解和肿瘤发生很重要,而MTR4是HCC的独立预后标志物。在这种情况下,4,19。
已知功能强大且普遍存在的癌蛋白c-Myc通过多种机制(例如调节包括PKM2 17在内的关键糖酵解基因的AS来促进癌症从氧化磷酸化到糖酵解的代谢转换)。据报道,c-Myc诱导PTBP1,hnRNPA1和hnRNPA2的表达,从而导致第10外显子的包涵和PKM2的产生12。我们的发现表明,c-Myc直接激活HCC细胞中MTR4的表达,从而提供了c-Myc如何调节HCC中糖酵解基因的AS从而诱导癌症代谢的另一种机制。考虑到c-Myc在大多数人类癌症中过表达并促进其肿瘤发生,但是MTR4在有限类型的人类癌症中过表达,我们预测c-Myc对于激活MTR4和其他HCC的转录是必要但不足的需要特定的转录因子才能与c-Myc合作激活HCC中的MTR4转录。
方法
患者和组织样本
在获得足够的知情同意后,从南方医科大学南方医院的77例接受了肝癌根治性切除术的患者中获得了肝细胞癌(HCC)组织和邻近的正常组织(ANT,超出肿瘤边缘至少2 cm),中国广州,2010年11月至2015年5月。通过门诊随访或电话访谈定期评估患者的生存分析。所有患者均满足以下入选标准:术前未接受任何其他治疗;确认手术切缘无残留癌组织。有关年龄,性别,肿瘤大小,肿瘤数目,肿瘤包膜,分化,AFP,肝硬化,MVI(微血管),PVTT(侵袭性门静脉肿瘤血栓形成),TNM分期的临床病理信息,并且BCLC阶段是可利用的。这项研究得到南方医科大学南方医院的IRB的批准,并且根据赫尔辛基宣言(第6版,2008年)进行。
立即将所有患者样品冷冻在液氮中,并保存在-80°C下直至提取RNA。为了进行mRNA表达水平和存活时间的相关性研究,根据制造商的说明,使用TRIzol试剂(Invitrogen,Carlsbad,CA)从组织中提取总RNA。使用PrimeScript cDNA试剂盒(Takara Bio,草津,滋贺,日本)合成cDNA。使用SYBR Premix Ex Taq(Takara Bio)进行实时定量PCR。18S RNA水平用作内部对照。PCR引物列于补充表 1中。用卡方检验评估基因表达与临床病理变量之间的相关性,包括年龄,性别,肿瘤大小,肿瘤数目,肿瘤包膜,分化,AFP,肝硬化,MVI,PVTT,TNM分期和BCLC分期。根据MTR4水平将患者分为两组:HCC中MTR4表达最高的前25个百分位的患者和其余患者。术后生存率采用Kaplan-Meier方法进行分析,生存率差异采用对数秩检验进行评估。单变量和多变量分析基于Cox比例风险回归模型。所有统计分析均使用IBM SPSS Statistics 20.0(IBM,IL,美国)进行。计算两侧的p值,并p <0.05被认为是统计学上显着的结果。
细胞系,病毒载体,构建体和转染
293 FT细胞系购自Thermo Fisher Scientific(马萨诸塞州沃尔瑟姆)。其他细胞系获自ATCC(美国美国典型培养物保藏中心),并保存在我们的实验室中。将所有细胞系在含有10%胎牛血清(FBS)(Gibco,澳大利亚)的DMEM(Invitrogen,Carlsbad,CA)中于5%CO 2在37°C下培养。为了建立具有稳定沉默基因的细胞,根据Addgene的pLKO.1协议,使用了通过pLKO.1 puro载体表达短发夹RNA(shRNA)的慢病毒(Addgene#8453)。港铁4shRNA靶序列是GGAAGGATTTCCGATGGATTT。根据Addgene的pLKO.1规程设计,退火并插入shRNA寡聚体到PLKO.1 puro中。慢病毒是通过将pLKO.1载体共转染,将质粒psPAX2(Addgene质粒12260,Addgene,Cambridge,MA)和包膜质粒pMD2.G(Addgene质粒12259)包装到HEK293FT细胞中而产生的。对于强力霉素(Doxy)诱导的MTR4敲低,使用了tet-pLKO-puro载体(Addgene#21915)和Fuw-M2rtTA(Addgene 20342)。感染后72小时,通过以2000 rpm离心10分钟去除培养基中的细胞碎片,并通过Lenti-X浓缩器(Clonetech,Mountain View,CA)浓缩慢病毒。病毒感染前一天,将细胞以1×10 5的密度接种到六孔板中 细胞/孔并在5μg/ ml聚丙烯存在下转导72小时。选择细胞对嘌呤霉素抗性(2μg/ ml)7天。为了过表达Glut1,将Glut1 cDNA的全长插入通过Xba1消化线性化的pLenti-CMV GFP载体(Addgene 17448)中。
使用小基因进行AS分析
由于技术问题,PKM通过将内含子8和内含子11之间的两个片段结合在一起,构建小基因,然后以H1 / H9 gDNA为模板进行PCR扩增,然后将其亚克隆到pLenti-CMV质粒中。通过测序确认在每个步骤中构建的所有质粒。使用一对引物hPKM-in8-F01和hPKM-R02通过PCR获得第一片段(3654 bp),并使用一对引物hPKM-in8-gF和hPKM-R02-g通过第二轮PCR进行吉布森装配修饰。修饰的片段通过线性化并通过BamHI / Sall酶切的载体骨架质粒pLenti-CMV GFP载体(Addgene 17448)应用于Gibson组装体(NEBuilder HiFi DNA Assembly Master Mix)。使用引物对hPKM-F02和hPKM-in11-R通过PCR获得第二个片段,并使用另一个引物对hPKM-Frag2-gF2和hPKM-gR通过PCR修饰得到第二个片段,并通过Sall消化和rSAP介导的脱磷酸作用将pLenti-CMV载体(包括第一个片段)用于吉布森装配。通过定点诱变将MTR4结合基序中的点突变引入到小基因中。对于poly-A尾部的突变,使用两个引物对(PKM-m1766-F,PKM-m1766-R; PKM-m1906-F,PKM-m1906-R)扩增cDNA产物,并与甲基化-依赖性核酸内切酶DpnI去除亲本模板。为了突变短的MTR4结合基序,使用了另外两个引物对(PKM-m1708-F,PKM-m1708-R; PKM-m1906-F,PKM-m1906-R)。使用两个引物对(PKM-m1766-F,PKM-m1766-R; PKM-m1906-F,PKM-m1906-R)扩增cDNA产物,然后将其与甲基化依赖性核酸内切酶DpnI孵育以去除亲本模板。为了突变短的MTR4结合基序,使用了另外两个引物对(PKM-m1708-F,PKM-m1708-R; PKM-m1906-F,PKM-m1906-R)。使用两个引物对(PKM-m1766-F,PKM-m1766-R; PKM-m1906-F,PKM-m1906-R)扩增cDNA产物,然后将其与甲基化依赖性核酸内切酶DpnI孵育以去除亲本模板。为了突变短的MTR4结合基序,使用了另外两个引物对(PKM-m1708-F,PKM-m1708-R; PKM-m1906-F,PKM-m1906-R)。
通过使用对内含子2到内含子8有特异性的引物对(hGlut1-in2-F,hGlut1-in8-R),通过PCR生成Glut1小基因。使用第二对引物hGlut1-in2-gF和hGlut1-in8-gR,通过第二轮PCR对所得产物进行Gibson组装修饰,并应用于Gibson组装。GLUT1小基因中定点诱变的引物是Glut1m-F和Glut1m-R。用于分析PKM和GLUT1中的替代剪接基因,用表达所示小基因的质粒转染PLC / PRF / 5细胞。48小时后,提取RNA,反转录,并用于PCR检测其他剪接形式。为了区分小基因衍生的转录本和内源性转录本,使用了对质粒骨架特异的反向引物。补充表2提供了用于小基因分析的所有引物的序列 。
细胞生长测定
对于克隆形成测定,通过胰蛋白酶消化制备单细胞悬液,并将指定的细胞系以每孔500个细胞的密度播种到6孔板上。培养2周后,将菌落用结晶紫染色并计数。使用96孔板进行细胞计数试剂盒8(CCK8)分析。简短地说,将细胞以3-5×10 3个细胞/孔的密度播种,并培养24-96小时。将CCK-8溶液添加到每个孔中,并孵育60分钟,用酶标仪在450nm处测量板。
动物实验
所有动物工作均得到南方医科大学和加利福尼亚大学圣地亚哥分校的机构动物护理和使用委员会(IACUC)的批准。为了分析NODSCID小鼠的肿瘤生长,收集细胞,将其重悬于以1:1比例与Matrigel混合的0.2 ml无血清DMEM中,然后皮下注射到NODSCID小鼠的左右侧面(每个5×10 6细胞) 。为了评估MTR4耗竭对致瘤性的影响,将直径约0.5 cm的已建立肿瘤的小鼠随机分为两组。随后,每天腹膜内(ip)注射Doxy(20 mg / kg体重)。对照组注射等体积的PBS。另外,向doxy治疗组连续提供含doxy的饮用水(2mg / L)。
细胞外酸化率(ECAR)
使用Seahorse XFe96细胞外通量分析仪(加利福尼亚州圣克拉拉,安捷伦)进行糖酵解压力测试(103020-100,安捷伦,加利福尼亚州圣克拉拉)。总共将2×10 4个细胞接种到XF96细胞培养微孔板(102416-100,安捷伦,加利福尼亚州圣克拉拉)的孔中,并孵育过夜。在无CO 2的情况下,在补充有2 mM谷氨酰胺的无缓冲XF分析培养基中平衡板1小时。通过连续添加葡萄糖(10 mM),寡霉素(1μM)和2-脱氧葡萄糖(2-DG,50 mM)来建立糖酵解(ECAR对葡萄糖-ECAR的反应)来测定细胞外酸化率(ECAR)葡萄糖注射前),糖酵解能力(对寡霉素的ECAR-葡萄糖注射前的ECAR),糖酵解储备(糖酵解能力-糖酵解)和非糖酵解酸化(葡萄糖注射前的ECAR)。每个标绘值是至少3个一式三份孔的平均值,并根据基线ECAR和总蛋白水平进行标准化。数据表示为平均值±SD,并通过两步方差分析与Tukey多重比较检验计算出显着性。
耗氧率(OCR)
使用Seahorse XFe96细胞外通量分析仪(加利福尼亚州圣克拉拉,安捷伦)测量细胞Mito应力测试(103015-100,安捷伦,加利福尼亚州圣克拉拉)。总共将2×10 4个细胞接种到XF96细胞培养微孔板(102416-100,安捷伦,圣克拉拉,加利福尼亚州)的每个孔中,并孵育过夜。在无CO 2的情况下,将板在补充有10 mM葡萄糖和2 mM谷氨酰胺的无缓冲XF分析培养基中平衡1小时。通过连续添加寡霉素(1μM),羰基氰化物4-(三氟甲氧基)苯hydr(FCCP,1.5μM)和鱼藤酮/抗霉素A(Rot / AA,0.5μM)来询问耗氧率(OCR),以建立基础呼吸(在寡霉素注射之前的OCR –响应Rot / AA的OCR),ATP的产生(在寡霉素注射之前的OCR –响应寡霉素的OCR),最大呼吸(响应FCCP的OCR −响应Rot / AA的OCR),备用容量(最大呼吸-基础呼吸)和质子泄漏(响应寡核苷酸的OCR-响应Rot / AA的OCR)。每个标绘的值是至少三个重复的孔的平均值,并已标准化为基线OCR和总蛋白水平。数据表示为均值±SD,并通过Tukey多重比较测试的双向ANOVA计算得出显着性。
免疫印迹和免疫共沉淀
样品在8-15%SDS PAGE上分离并转移到硝酸纤维素膜上。将印迹在含有一抗的封闭缓冲液(含0.05%Tween 20的PBS中的5%脱脂奶)中孵育。用封闭缓冲液洗涤3次后,用辣根过氧化物酶偶联的二抗探测印迹,并用Supersignal West Pico或Dura(Thermo Fisher Scientific)显影。使用了以下抗体:兔多克隆抗Myc抗体(13987; Cell Signaling),兔多克隆抗MTR4抗体(ab70551; Abcam),抗Glut1抗体(ab150299; Abcam),小鼠单克隆抗α-微管蛋白抗体( T5168; Sigma-Aldrich),抗兔IgG,HRP连接的抗体(7074S;细胞信号转导),抗小鼠IgG抗体,HRP连接的抗体(7076S;细胞信号转导)。对于共同免疫沉淀(Co-IP),将细胞裂解物在IP裂解缓冲液(87787,Thermo)中与Halt蛋白酶和磷酸酶抑制剂混合物(78430,Thermo)在冰上制备30分钟。以12,000离心后× g在4°C下放置10分钟,将上清液用抗hnRNPA1(03-204,Millipore)或抗PTBP1(MABE-986,Millipore)抗体以及磁性蛋白A / G磁珠(Pierce)免疫沉淀2 h在4°C下。使用Image Lab(BioRad)定量蛋白质条带的强度。补充信息中的补充图7提供了所有未经裁剪和未经处理的蛋白质印迹扫描 。
定量PCR分析
根据制造商的说明,使用RNeasy Mini Kit(Invitrogen)从细胞中提取总RNA。将细胞直接收集到RLT裂解缓冲液中,并根据制造商的说明使用QIAshredder(Qiagen)进行均质化。用RNeasy Mini试剂盒柱纯化RNA,并用无RNase的DNase Kit(Qiagen)除去基因组DNA。TRIzol试剂(Thermo Scientific)用于从人类患者组织中提取RNA。用电动匀浆器将0.5 mg肝组织在1 ml TRIzol试剂中匀浆,然后与0.2 ml氯仿剧烈混合(Sigma-Aldrich)。在室温下孵育3分钟后,将混合物以10,000× g离心 15分钟 将RNA级分与相同体积的100%乙醇混合,并用RNeasy柱纯化。使用高容量cDNA逆转录酶试剂盒(Applied Biosystems)按照制造商的说明进行总RNA的cDNA合成。使用FastStart Universal SYBR Green Master(Takara),通过ABI Prism 7000(Applied Biosystems)进行实时定量PCR。PCR条件如下:在95°C下2分钟,在95°C下5 s的40个循环,在60°C下34 s。通过一式三份的反应确定每个基因的平均Ct值,并用β-肌动蛋白的mRNA水平或18 s进行标准化。补充表1中提供了用于qPCR分析的所有引物的序列 。
RNA免疫沉淀和测序(RIP-seq)
根据制造商的说明,使用Magna RIP RNA结合蛋白免疫沉淀试剂盒(Millipore;美国)进行RNA免疫沉淀(RIP)。用含有蛋白酶抑制剂鸡尾酒(Thermo Fisher,美国)和RNase抑制剂(Invitrogen,美国)的RIP裂解缓冲液裂解过表达MTR4-HA的细胞。要获得表达MTR4-HA的质粒,需要全长MTR4用标记有HA序列的引物(TACCCATACGATGTTCCAGATTACGCT)扩增cDNA,并将其插入通过KpnI / NotI消化线性化的pcDNA3载体中。收集核沉淀并通过超声处理将其裂解。将核裂解液与抗HA抗体或对照IgG在4°C下孵育过夜,然后与A / G Dynabeads蛋白(Thermo Fisher,美国)孵育2 h。剧烈洗涤后,将沉淀重新悬浮在TRIzol(美国,Invitrogen)中。免疫沉淀的RNA通过RNAeasy Mini Kit(Invitrogen)纯化,逆转录并测序。用FasX-Toolkit检查所有测序读物,并用cutadapter除去衔接子。使用TopHat v2.0.13将已处理的读段映射到hg19全基因组。高峰通话是使用RIPSeeker执行的。峰值标注是用荷马完成的,以及使用MEME-ChIP识别的与MTR4结合的基序。为了评估MTR4结合基序对糖酵解基因RNA与RNA结合蛋白之间相互作用的影响,将表达WT小基因或具有MTR4结合基序突变的小基因的细胞衍生的cDNA用于PCR,使用对该小基因特异的反向引物向量。补充表中提供了用于RIP-seq分析的所有引物的序列 3。
肿瘤样本的免疫组织化学(IHC)分析
肿瘤样品在0.1 M磷酸盐缓冲液中用4%多聚甲醛固定24小时,包埋在石蜡中,切成4μm切片,并安装在载玻片上。脱蜡后,将载玻片用3%的甲醇中的过氧化氢处理,并用生物素封闭试剂盒(DAKO,德国)封闭,与Ki67单克隆抗体(1:50,BD Biosciences,USA)在4℃的潮湿室内孵育过夜。 ℃。用PBS洗涤3次后,将载玻片与生物素化的山羊抗小鼠抗体孵育1小时。用DAKO液体3,3'-二氨基联苯胺四盐酸盐(DAB)将载玻片染色,然后用Mayer's苏木精复染并在显微镜下检查。通过半定量IHC检测确定Ki67的蛋白水平。
细胞周期分析
将细胞以1×10 6个 细胞/孔的密度接种到6孔板上,并用MMC(5 µg / ml)处理12 h。将细胞用冷的70%乙醇在4°C下固定24小时,然后与0.5 mg / ml的碘化丙啶(PI)以及0.1 mg / ml的RNase A(MultiSicences,中国)一起孵育。使用流式细胞仪(FACScan,BD Biosciences)进行细胞周期分析,并使用ModFIt LTV4.1.7软件(BD Biosciences)进行分析。门禁细胞碎片和死细胞用于细胞周期分析。
染色质免疫沉淀(ChIP)
用1%甲醛将蛋白质与DNA交联后,提取细胞核,并用微球菌核酸酶消化,产生〜150–600 bp的DNA片段。在4°C下用抗c-Myc抗体免疫沉淀过夜后,将免疫沉淀的染色质的交联反转,并通过qPCR测量免疫沉淀中的DNA量。
选择性剪接的mRNA亚型的RNA序列分析
我们使用带有默认参数的软件hisat2(v2.1.0)将RNA-seq读图映射到人类基因组(H. sapiens,GRCh38)和转录组(Ensembl,版本84)。使用rMATS(rMATS)分析了与AS模式的所有五种基本类型相对应的两个样本组(KD与对照)之间的差异性可变剪接(AS)事件。我们使用-c 0.0001参数运行rMATS,然后使用FDR <5%和| IncLevelDifference |的临界值检测到重要的剪接事件。≥5%,导致发生1713 SE,131 A5SS,192 A3SS,307 MXE和103 RI差分AS事件。
量化和统计分析
Kaplan-Meier生存图的统计学意义通过对数秩分析确定。单变量和多变量分析基于Cox比例风险回归模型。通过重复测量方差分析测试肿瘤生长率的统计学显着性。为了比较两组之间的肿瘤重量,使用了非参数Mann-Whitney检验。对于其他实验,通过t检验和方差分析确定显着差异。星号表示显着差异:ns,不显着。我们假设具有少量数据的正态分布。使用IBM SPSS Statistics 20.0(IBM)对患者的生存情况进行所有统计分析。使用PRISM进行了其他分析。ñ在图例中指出。没有使用统计方法来确定样本大小,盲目化和随机化。
生物材料的可获得性
通过将请求发送给相应的作者,可以获得所有生物材料。
报告摘要
链接到本文的《自然研究报告摘要》中提供了有关研究设计的更多信息 。
资料可用性
图2a和补充图 3中的RNA-seq数据 可在NCBI基因表达综合(GEO)中获得,登录号为GSE129263。图6a和补充图 5中的RIP-seq数据 可在GSE129261下的NCBI基因表达综合总线(GEO)中获得。从Gene Expression Omnibus(GEO)数据库以登录号GSE14520获得了225个HCC组织和220个非癌性肝组织的转录数据集。补充图7中包括未裁剪和未处理的Western blot和凝胶的图像。 。无花果的源数据 图1c-f,1h-i,1k-1,图2b-f,图3b-e,提供图4c,4e,4h-k,图5a-b,图6b,补充图1,补充图2b,c,eh,k,补充图5a-c作为源数据文件。
|
|